Resistencias del VIH-1 y el laboratorio clínico
J. Córdoba, R.J. Esteban, R. Garijo, J.M. Molina y M. Gobernado Servicio de Microbiología, Hospital Universitario La Fe, Avda. Campanar 21, 46009 Valencia.
|
ESTRUCTURA Y CICLO DE REPLICACIÓNDEL VIH-1 En 1983, el grupo de Montagnier (1) aisló un virus que se denominó virus asociado a la linfadenopatía (LAV), y al año siguiente el grupo de Gallo (2) aisló otro virus al que denominaron virus linfotrópico humano III (HTLV-III). Posteriormente, estudios de secuenciación del genoma de ambos virus demostraron que era el mismo, admitiéndose internacionalmente una nueva denominación, la de virus de la inmunodeficiencia humana (VIH), para el agente causal del síndrome de la inmunodeficiencia adquirida (sida) (3). Hasta nuestros días se han identificado dos tipos de VIH: el 1, causante de la mayor parte de las infecciones en los países desarrollados, y el 2, aislado por primera vez en 1986 (4), localizado fundamentalmente en África occidental y de menor patogenicidad. El VIH es un retrovirus perteneciente a la subfamilia Lentiviridae. Los lentivirus se han aislado en diferentes especies de mamíferos superiores y se caracterizan por producir enfermedades de evolución lenta que pueden dar lugar a tres tipos de síndromes, a saber, neurológicos, autoinmunitarios y causantes de inmunodeficiencia. En cuanto a su estructura, los viriones del VIH son partículas esféricas de entre 80 y 100 nanómetros en las que podríamos distinguir tres capas concéntricas: una envoltura lipidoproteica, una nucleocápside icosaédrica central y en el interior de ésta tanto el material genético como las enzimas necesarias para completar su ciclo vital (transcriptasa inversa, integrasa y proteasa) (Fig. 1). Al igual que los demás retrovirus, su genoma es un RNA de cadena única constituido por dos hebras idénticas, de polaridad positiva. El genoma del VIH tiene una longitud de 9800 pares de bases, presentando tres genes estructurales y al menos seis genes reguladores, que en el estado de provirus están limitados por unas secuencias repetidas (LTR) que facilitan su integración en el genoma de la célula huésped y que contienen los elementos reguladores del inicio de la transcripción viral (5). 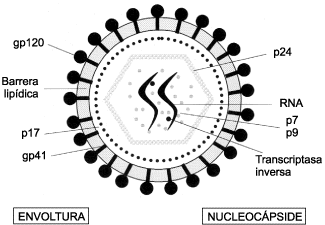 Figura 1. Estructura del VIH. En el ciclo biológico del VIH (6) podemos distinguir dos fases bien definidas: una temprana, en la cual se consuma la integración del DNA proviral en el genoma de la célula huésped, y una fase tardía en que se activan los procesos de transcripción viral dando lugar a la formación y diseminación de gran número de viriones infectantes. En la mayoría de las infecciones causadas por lentivirus el tropismo es exclusivamente macrofágico (7, 8), lo que da lugar a una replicación viral de menor intensidad y agresividad, generando habitualmente cuadros clínicos neurológicos sin inmunodeficiencia asociada. Por el contrario, la cinética de replicación del VIH es muy rápida, habiéndose calculado que una persona infectada puede generar alrededor de 109 a 1010 viriones por día (9, 10). Esto supone que la vida media plasmática de un virión es inferior a las seis horas y la de un linfocito infectado que replica activamente el VIH (104 a 1010 partículas virales) es de 1,2 días. La capacidad del VIH para producir una gran inmunodeficiencia radica, a diferencia del resto de los lentivirus, en su capacidad de infectar una célula central en la regulación de la respuesta inmunitaria, los linfocitos CD4+. Además, su capacidad de adaptación al entorno celular de estos linfocitos le permite a su vez una situación de latencia proviral y de replicación acelerada, con la consiguiente destrucción del sistema inmunitario (11). Cuando estudiamos la carga viral, durante la evolución de un paciente infectado por el VIH se pueden establecer tres periodos (11). El primero, el periodo de primoinfección, que se desarrolla en las primeras semanas tras el contacto con el virus y que se caracteriza, independientemente de la existencia o no de síntomas, por una carga viral plasmática muy elevada (>750.000 copias/ml). Posteriormente, en el transcurso de varias semanas se produce una reducción significativa (2-4 logaritmos) de la carga viral hasta alcanzar un nivel relativamente estable, con el que pasa a la segunda fase llamada de replicación crónica. Se inicia así un periodo asintomático de la enfermedad cuya carga viral estable se considera como el principal factor pronóstico de evolución a sida. Finalmente, en un momento dado de la evolución, el paciente entra en la fase acelerada de replicación viral que desencadena la fase final de la enfermedad. En este momento, el aumento de la carga viral suele preceder a una caída de la cifra de linfocitos CD4+, que constituye un signo de mal pronóstico en la evolución (Fig. 2). MUTACIONES Y RESISTENCIA Durante todas las fases descritas el VIH tiene que evitar su eliminación por parte de la respuesta inmunitaria de la persona infectada, para lo cual desarrolla fundamentalmente cuatro mecanismos de escape: a) su capacidad para generar una gran variabilidad en las proteínas del virus aprovechando su elevada tasa (10-3 a 10-4) de error de la transcriptasa inversa; b) la existencia en su ciclo biológico de fases de latencia y de reactivación; c) la adaptación a una serie de receptores celulares que le permiten infectar y destruir el sistema inmunitario; y d) la capacidad para acantonarse en reservorios de difícil acceso para la respuesta inmunitaria. Todos estos mecanismos facilitan la existencia de una gran variabilidad en las poblaciones del VIH-1, a la vez que le otorgan una extraordinaria capacidad de adaptación a la presión del medio ambiente, ya sea realizada por el sistema inmunitario o con la administración de antirretrovirales. Dentro de la heterogénea población viral que infecta a un individuo podemos distinguir distintas cuasiespecies, que se pueden definir como distribuciones dinámicas de genomas virales no idénticos, pero genéticamente muy cercanos, que se hallan sometidos a un continuo proceso de variación, competición y selección (12). La probabilidad de encontrar una determinada mutación entre las cuasiespecies que infectan a un paciente depende de la ventaja adaptativa (fitness) o desventaja que su existencia confiera al virus (13). La extraordinaria tasa de replicación del VIH-1 da lugar a la continua producción de variantes con mutaciones, que no llegan a ser mayoritarias en la población de cuasiespecies hasta que no existe una presión farmacológica. Cuando se administra un antirretroviral se produce una selección de las variantes del virus preexistentes que poseen una o varias mutaciones que confieren resistencia al fármaco, pasando a ser la población predominante y quedando suprimidas las variantes sensibles. 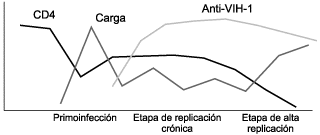 Figura 2. Evolución de la infección por el VIH. Llegados a este punto parece razonable señalar una serie de conceptos que en ocasiones pueden inducir a confusión. Así, utilizaremos el término "resistencia primaria" para designar las cepas resistentes en pacientes que nunca han recibido tratamiento antirretroviral, y "resistencia secundaria" para designar las cepas que aparecen como consecuencia de la selección por tratamientos subóptimos. Los conceptos de resistencia primaria y secundaria hay que diferenciarlos de los de resistencia genotípica, conferida por mutaciones primarias, secundarias y compensatorias. Por mutaciones primarias se entienden aquellos cambios del genoma viral que codifican resistencia, en general de alto grado, y que normalmente son las primeras que se seleccionan. Las mutaciones secundarias suelen seleccionarse tras las primarias si persiste una presión selectiva (tratamiento subóptimo), y en general aumentan el grado de resistencia. Las mutaciones compensatorias suelen ser las más tardías en aparecer y pueden permitir al virus recuperar parte de la capacidad de replicación o fitness que podía haber perdido por la presencia de mutaciones primarias o secundarias. Por último, se denomina "resistencia fenotípica" a la capacidad del virus de requerir una mayor concentración de un determinado fármaco para poder inhibir su replicación en un sistema de laboratorio. Suele estar causada por la existencia de una o varias mutaciones. Cuando se precisan concentraciones entre 4 y 10 veces por encima de las necesarias para inhibir la cepa de referencia, suele hablarse de resistencia intermedia; cuando se precisan concentraciones superiores a 10 o más veces se habla de resistencia de alto grado (14). TERAPIA ANTIRRETROVIRALY SELECCIÓN DE MUTANTES RESISTENTES Para controlar la infección por el VIH-1, actualmente contamos con unos diez inhibidores de la transcriptasa inversa y cinco inhibidores de la proteasa. Estos fármacos, tanto en monoterapia como en combinación, suelen producir en los pacientes un descenso de la carga viral del VIH-1, pero la eficacia de la mayoría de los regímenes terapéuticos sólo es parcial. El descenso del número de células infectadas y de la carga viral suele ir acompañado de un aumento en el número de CD4+ y de células vírgenes, y también de un aumento transitorio, poco importante, de células T memoria (15, 16). Incluso tras varios meses de la aplicación de una terapia combinada muy agresiva (HAART: Highly Active Antiretroviral Therapy) el número de células funcionales del sistema inmunitario dista mucho de alcanzar valores normales. Se ha comprobado que a las pocas semanas de la instauración de terapias agresivas se ralentiza el descenso de la carga viral, lo que sugiere la existencia de reservorios estables de células infectadas. Estos reservorios, poco accesibles para los fármacos aun con terapias agresivas, constituyen la principal dificultad para la erradicación del virus (17). De este modo, la incompleta supresión de la replicación viral genera la aparición de cepas con mutaciones que confieren resistencia a uno o más fármacos, siendo ésta una de las principales causas del fracaso terapéutico. La aparición de cepas mutantes resistentes al tratamiento es un proceso en el cual las distintas variantes coexisten en un mismo individuo. Se han detectado mutaciones que confieren resistencia a diversos inhibidores de la transcriptasa inversa y de la proteasa. No obstante, se admite que en la fase previa a la terapia antirretroviral la baja o moderada prevalencia de las cuasiespecies mutantes responde a su menor capacidad replicativa respecto a las variantes silvestres. En el seguimiento clínico de los pacientes, la carga viral plasmática permite constatar el inicio de un fracaso terapéutico. La detección de variantes resistentes nos puede permitir correlacionar dicho fracaso con los distintos fármacos administrados y optimizar la terapia. Las técnicas para la determinación de las resistencias del VIH-1 a los distintos antirretrovirales se pueden clasificar en dos grandes grupos: genotípicas, que detectan mutaciones en el genoma del virus, y fenotípicas, que determinan la sensibilidad del virus a distintos fármacos. En principio las técnicas genotípicas se caracterizan por su sensibilidad, ser menos laboriosas, más rápidas y de coste muy inferior a las fenotípicas. No obstante, en el momento actual no parece existir un consenso general para el uso de la información que generan las pruebas genotípicas como factor predictivo de respuesta al tratamiento antirretroviral. La existencia de cuasiespecies virales no detectadas, el desconocimiento del efecto de algunas mutaciones y sus resistencias cruzadas, así como de la variable respuesta de los pacientes a los fármacos, avalan la precaución de la comunidad científica (18). Las técnicas fenotípicas cuantifican el grado de sensibilidad a un fármaco específico, pero son más laboriosas y complejas, no detectan variantes minoritarias, pueden seleccionar las cepas mejor adaptadas al cultivo celular y la significación clínica de sus resultados está por definir. TÉCNICAS GENOTÍPICAS Las técnicas genotípicas se utilizan para establecer la presencia de mutaciones genómicas que generen cambios en la secuencia de aminoácidos y, por lo tanto, en las proteínas del VIH-1. Se basan en la amplificación por PCR de aquellas regiones implicadas en el desarrollo de resistencias, habitualmente el gen de la transcriptasa inversa y el gen de la proteasa, para el posterior análisis de su secuencia de nucleótidos. No es necesario el aislamiento del virus, permitiendo además el procesamiento simultáneo de distintas muestras en las que se pueden detectar diversas mutaciones representativas del conjunto de cuasiespecies del paciente en estudio. La amplificación por PCR del genoma del VIH-1 en general, y de sus cuasiespecies en particular, se ve facilitada por una carga viral superior a 1000 copias de RNA del VIH-1 por mililitro de plasma y por la presencia de una determinada cuasiespecie en un porcentaje superior al 10% a 20%. Entre las distintas técnicas genotípicas actualmente en uso destacaríamos las siguientes: – Ensayos LiPA HIV-1 RT y LiPA HIV-1 P. – Secuenciación de ácidos nucleicos. – Afimetrix. LiPA HIV-1 RT y LiPA HIV-1 P El ensayo LiPA HIV-1 RT permite la detección simultánea de tipos salvajes, mutaciones o polimorfismos en los codones 41, 69, 70, 74, 184, 214 y 215 del gen de la transcriptasa inversa del VIH-1. Se basa en el principio de hibridación inversa tras la amplificación por PCR del gen de la transcriptasa inversa, donde el DNA biotinilado es sometido a hibridación con oligonucleótidos específicos inmovilizados como líneas paralelas en una tira de nitrocelulosa. El resultado de la hibridación se evidencia como bandas coloreadas mediante el uso de fosfatasa alcalina marcada con estreptavidina y el cromógeno BCIP/NBT. La interpretación de los resultados de la hibridación anterior se realiza comparando la reactividad frente a las diferentes sondas con el patrón incluido en el ensayo (19). En el laboratorio, esta técnica permite el análisis rápido y simultáneo del gen de la transcriptasa inversa en un número importante de muestras (20). En el momento actual se esta ensayando el LiPA HIV-1 P para la detección de las mutaciones que confieren resistencia a los fármacos inhibidores de la proteasa. El fundamento de esta técnica es semejante al descrito anteriormente, pero utilizando iniciadores y sondas derivados del gen de la proteasa del VIH-1, siendo llamativa la no inclusión de sondas para la detección de mutaciones en el codón 90. Ambas pruebas presentan una gran sensibilidad para detectar variantes minoritarias, pero la abundancia de polimorfismos en algunas muestras puede dificultar la hibridación y, por consiguiente, la obtención de resultados. Como en otras pruebas genotípicas, la interpretación de los resultados es laboriosa y complicada, especialmente en pacientes politratados en los cuales los patrones de resistencia suelen ser complejos. Secuenciación de ácidos nucleicos La secuenciación es el método genotípico de referencia, que proporciona información de todos los nucleótidos de la región en estudio. Para la determinación de las resistencias del VIH-1, la práctica más extendida es la secuenciación cíclica a partir de productos de PCR obtenidos de la amplificación de los genes de la transcriptasa inversa y de la proteasa del virus. Los productos secuenciados mediante el uso de iniciadores o didesoxinucleótidos marcados se detectan en el curso de una electroforesis en equipos automáticos. Las secuencias de nucleótidos obtenidas deben ser comparadas con las secuencias de referencia del VIH-1 para determinar la presencia de las distintas mutaciones. En nuestro país actualmente se encuentran disponibles dos equipos comerciales para la detección de las mutaciones del VIH-1 basados en la secuenciación cíclica (PE Biosystems y Visible Genetics). Los principales obstáculos para la incorporación de estas técnicas en los laboratorios son, por un lado, su elevado coste económico, y por otro la necesidad de contar con personal especializado en biología molecular. No obstante, el coste económico queda minimizado ante la posibilidad de optimizar las terapias antirretrovirales utilizando la información que proporcionan estas pruebas genotípicas. Afimetrix Este método consiste en la hibridación de pequeños fragmentos de RNA obtenidos del aislamiento por PCR y la transcripción in vitro. Los fragmentos, marcados con biotina, se hibridan a un microchip en el cual están incluidos una gran cantidad de oligonucleótidos solapantes que permiten la identificación del nucleótido presente en cada posición. Esta técnica permite procesar de manera rápida un número elevado de muestras. Aunque parece una tecnología futurista, presenta los mismos problemas que el resto de los ensayos genotípicos debidos a la elevada heterogeneidad del virus. La existencia de polimorfismos puede alterar la hibridación de los fragmentos y hacer difícil la detección de ciertas mutaciones. Además, la presencia de deleciones e inserciones puede plantear problemas en la detección de la secuencia. El Afimetrix ha sido empleado con éxito para estudiar la heterogeneidad y la presencia de mutaciones en el gen de la proteasa del VIH-1. Los estudios que comparan los resultados obtenidos por este método y los de secuenciación automática demuestran que ambas técnicas son equiparables (21). Por otro lado, el Afimetrix no está diseñado para identificar mezclas de poblaciones virales. TÉCNICAS FENOTÍPICAS Las técnicas fenotípicas permiten conocer si una determinada cepa del VIH-1 es sensible o resistente a un fármaco, teniendo en cuenta su capacidad de replicación ante concentraciones decrecientes del fármaco in vitro (14). El resultado que se obtiene se conoce como concentración inhibitoria del 50% (CI50) o del 90% (CI90), lo que indica la concentración de fármaco necesaria para reducir la capacidad de replicación del VIH-1 en un 50% o en un 90%, respectivamente. Estas técnicas requieren el aislamiento y la cuantificación del virus en sangre periférica. Los aislamientos son cultivados con células mononucleares de sangre periférica (CMSP) que se obtienen de donantes sanos y que son estimuladas en presencia de diluciones seriadas de un determinado fármaco o de una combinación de ellos. La sensibilidad relativa del virus a un fármaco se obtiene en función de la menor concentración de fármaco capaz de suprimir la replicación viral determinada por la cuantificación del antígeno p24, lisis celular o inducción de sincitios, según sea el sistema de cultivo celular utilizado (22-24). La determinación fenotípica de la sensibilidad del VIH-1 se considera actualmente como el patrón de referencia, pero también cuenta con importantes inconvenientes: – Laboriosidad. – Coste de instalaciones y de personal. – Falta de estandarización. – Necesidad de contar con instalaciones de seguridad biológica de nivel 3. – Está por definir la importancia clínica de los valores de CI50 y CI90. – No detectan el desarrollo de resistencias hasta que no se incrementa la CI50, por lo que la determinación genotípica suele ser más precoz. – Al igual que las técnicas genotípicas, sólo se obtienen resultados con muestras de pacientes con carga viral superior a 1000 copias de RNA del VIH-1 por mililitro de plasma, con lo cual tampoco se detectan variantes resistentes en baja proporción. – Posibilidad de seleccionar las cepas mejor adaptadas al cultivo y que podrían no ser representativas del conjunto de cuasiespecies existentes en el paciente. Para intentar evitar estos importantes inconvenientes se han desarrollado métodos de laboratorio que combinan las técnicas fenotípicas y genotípicas (25-27). En estos métodos combinados se comienza extrayendo RNA del VIH-1 del plasma de pacientes con carga viral superior a 1000 copias/ml, y tras transcripción inversa se realiza una amplificación de los genes de la transcriptasa inversa y de la proteasa del virus. Los productos amplificados son clonados en un vector VIH-1 plasmídico, en el cual los genes de la transcriptasa inversa y de la proteasa han sido delecionados previamente. Como resultado se obtiene un VIH-1 recombinante, con capacidad infecciosa, que será el utilizado para los ensayos fenotípicos. Los ensayos fenotípicos con virus recombinantes tienen tres ventajas: 1) Las cuasiespecies circulantes en los pacientes están mejor representadas. 2) No es necesario aislar el virus. 3) Se pueden automatizar. UTILIDAD DE LAS PRUEBAS PARA LA DETECCIÓN DE RESISTENCIA DEL VIH-1 En los últimos años estamos asistiendo a un esperanzador avance en el que, de manera progresiva, se están aprobando nuevos fármacos antirretrovirales. Desafortunadamente, nos enfrentamos a un microorganismo, el VIH-1, que cuenta con grandes recursos para evitar su erradicación. En efecto, su gran capacidad replicativa y por lo tanto su variabilidad genética le permiten un oculto proceso, paralelo a la aparición de nuevos fármacos, en el que se desarrollan resistencias directas a éstos y cruzadas a otros compuestos dentro de las distintas familias de antirretrovirales. Este desarrollo de resistencias es una causa frecuente e importante de fracaso terapéutico y presenta el riesgo de limitar los posibles regímenes alternativos. Como consecuencia, la comunidad científica muestra un creciente interés por el estudio de las resistencias del VIH-1, cuya caracterización y posible prevención repercutirá en la mejora de la atención a los pacientes. El desarrollo de métodos automatizados para los estudios genotípicos y fenotípicos nos permite considerar la utilidad de la información que generan dichas pruebas para el tratamiento de los pacientes con infección por el VIH. Tanto los ensayos genotípicos como los fenotípicos tienen sus ventajas y sus inconvenientes (14). Los ensayos fenotípicos tienen la ventaja de proporcionar una medida directa de la sensibilidad del VIH a los antirretrovirales. Sin embargo, presentan claras desventajas, entre las que cabe destacar: – Su disponibilidad restringida, por lo general limitada a los laboratorios de investigación. – Su complejidad técnica. – Costes elevados. – Gran lentitud en generar resultados. – Valores de CI50 que definen resistencias variables o aún no claramente establecidos para cada fármaco. – Selección de una población predominante, mejor adaptada al cultivo, no representativa de las poblaciones existentes en el paciente. Los ensayos genotípicos tienen la ventaja de ser técnicamente menos complejos y más rápidos. Además, en principio detectan antes las resistencias. Por el contrario, proporcionan una medida indirecta de la sensibilidad a los fármacos y también pueden seleccionar poblaciones virales no representativas de las existentes en los pacientes. Los métodos mixtos combinan las ventajas de ambos ensayos, es decir, la medida directa de la sensibilidad del virus como los fenotípicos y la rapidez de los genotípicos. La desventaja de los sistemas recombinantes es su elevado coste y su limitada disponibilidad en determinados laboratorios. Por otro lado, hay que tener en cuenta que el fracaso terapéutico no se debe exclusivamente a la aparición de resistencias (31, 32). El tipo de fármaco utilizado, la previa exposición a éste, la posibilidad de aparición de resistencias cruzadas, el incumplimiento del tratamiento, las alteraciones en el metabolismo intracelular y la farmacocinética del fármaco en los diferentes compartimientos del organismo tienen una importante influencia en la respuesta terapéutica. Estudios recientes han demostrado la utilidad de la secuenciación del genoma del VIH para guiar el cambio terapéutico en pacientes que no respondían al tratamiento antirretroviral (28, 29). Es importante destacar que cuando el genotipo se interpreta adecuadamente nos está dando también la información fenotípica. Asimismo, parece existir una buena correlación entre resistencias genotípicas y fenotípicas, lo que relegaría el estudio fenotípico a casos muy específicos en que exista un gran número de mutaciones. En tales casos no existen investigaciones que demuestren que la aplicación de los consejos derivados del estudio fenotípico presenten relevancia clínica adicional (30). En vista de todo lo anterior y a la hora de tener que decidir la incorporación de las técnicas descritas al laboratorio clínico, parece razonable dirigir nuestros esfuerzos hacia aquellas que tengan una mayor disponibilidad y que a su vez requieran menor tiempo de ejecución, complejidad técnica y coste económico. Obviamente, estas condiciones apuntan con claridad hacia las pruebas genotípicas, que sin duda están suponiendo una revolución en el enfoque del laboratorio clínico. Entre las pruebas genotípicas comercialmente disponibles también parece razonable inclinarse por las que, con costes semejantes, nos proporcionen más información. Así, las técnicas basadas en la secuenciación de ácidos nucleicos nos permiten conocer la secuencia completa de nucleótidos de los genes de la transcriptasa inversa y de la proteasa del VIH-1, mientras que las técnicas de hibridación sólo encuentran lo que buscan, es decir, únicamente nos informan de las posibles mutaciones que reconocen las sondas que incluyen (20). En resumen, las pruebas genotípicas son una realidad y deben ser incorporadas progresivamente en los laboratorios de diagnóstico clínico, lo cual permitirá a cada centro ir adquiriendo su propia experiencia en la utilidad de la información que proporcionan, teniendo en cuenta que el valor predictivo de estas pruebas se definirá mejor con la realización de estudios amplios enfocados al análisis de la importancia de las resistencias genotípicas en los diferentes regímenes terapéuticos. BIBLIOGRAFÍA
|