Elección y uso de las pruebas de detección de resistencias del VIH
J.M. Eiros, B. Hernández, C. Labayru
y R. Ortiz de Lejarazu
Servicio de Microbiología, Hospital
Universitario, Facultad de Medicina, Valladolid
|
INTRODUCCIÓN Desde el punto de vista conceptual, "elección" hace referencia a la acción de escoger o preferir para un fin determinado, y bajo el término "uso" puede englobarse la idea de hacer servir una cosa para algo. En este trabajo expondremos, de manera deliberadamente esquemática, algunas cuestiones relativas a cinco aspectos inherentes a esta cuestión. En primer lugar reflexionaremos acerca de las resistencias del virus de la inmunodeficiencia humana (VIH) al tratamiento antirretroviral. A continuación describiremos los tipos de técnicas comerciales disponibles para este fin, comentando sus ventajas e inconvenientes. En tercera instancia expondremos las recomendaciones al respecto, tanto de los organismos internacionales como de los nacionales. En cuarto lugar comentaremos determinados matices de su incorporación a la práctica asistencial. Y finalmente señalaremos algunas cuestiones pendientes. RESISTENCIAS DEL VIH AL TRATAMIENTO ANTIRRETROVIRAL Desde el punto de vista genérico los virus están provistos de una gran capacidad de adaptación a los cambios introducidos en su medio natural (1). En concreto, el VIH fundamenta esta cualidad en diversos mecanismos, entre los que cabe destacar cuatro: - La retrotranscriptasa carece de actividad exonucleasa 3'-> 5', que actúa como correctora de errores (1). - Tanto las proteínas estructurales como aquellas con actividad funcional poseen una notable plasticidad (2, 3). - El virus presenta una alta tasa de replicación, que permite generar del orden de 1010 "nuevos" viriones cada día (4). - Está bien documentado que en un determinado momento coexisten en cada persona infectada todas las posibles variantes del VIH ("cuasiespecies") (5, 6). Dada la notable heterogeneidad genética del VIH se producen procesos evolutivos, como la mutación que dará paso a la selección de cuasiespecies en virtud de su capacidad de supervivencia en un ambiente "hostil", y se producirá por tanto una selección de aquellas que presenten características de supervivencia más favorables en estas circunstancias (7, 8). Debido a que las mutaciones aparecen de forma espontánea y simplemente se seleccionan bajo la presión selectiva de los fármacos, actualmente el VIH tiene capacidad de desarrollar resistencias frente a todos los antirretrovirales disponibles, e incluso capacidad potencial frente a moléculas que están por diseñar (9-12). La resistencia en sentido amplio se define como cualquier cambio que mejore la replicación del VIH en presencia de un inhibidor (13). Es importante tener en cuenta que el concepto de resistencia es relativo, ya que si se parte de un inóculo lo suficientemente pequeño y se usa un fármaco en concentración suficiente, un virus resistente podría presentarse como sensible (14). En términos específicos, la resistencia del VIH al tratamiento antirretroviral consiste en un fenotipo alterado como resultado de un cambio en el genotipo viral que se puede detectar tanto in vitro como in vivo (15). Epidemiológicamente se puede hablar de resistencias primarias y secundarias (5, 16, 17). Hablamos de resistencias primarias cuando se detectan en virus de pacientes que no han sido tratados previamente, lo que implica que la infección se ha adquirido a partir de cepas de VIH resistentes. Por el contrario, diremos que se trata de resistencias secundarias cuando aparecen en la población viral de un paciente como consecuencia de la presión selectiva ejercida por la exposición a fármacos antirretrovirales. Tipos de mutaciones Existen dos tipos de mutaciones: primarias y secundarias. Se conocen como mutaciones primarias aquellas alteraciones en el material genómico que una vez expresadas darán lugar a cambios en el sitio activo de la enzima, dado que afectan a la afinidad de ésta por su sustrato. Habitualmente se seleccionan pronto en el curso del tratamiento por la presión selectiva ejercida por el fármaco, en un intento de evadir su acción inhibidora (18). Las mutaciones secundarias se van acumulando en el genoma viral que ya posee mutaciones primarias con la finalidad de restaurar la ventaja cinética que ha pagado la enzima por la mutación primaria. Son seleccionadas, pues, por la ventaja replicativa que confieren y la mejora de la afinidad por el sustrato natural de la enzima. Por sí mismas, estas mutaciones secundarias poseen un efecto mínimo o nulo en la magnitud de la resistencia al tratamiento antirretroviral (19). Justificación de la detección de resistencias del VIH Numerosos estudios retrospectivos realizados a lo largo de los años han demostrado la correlación entre la presencia de mutaciones y el fracaso terapéutico (20-27). Recientemente se han llevado a cabo estudios prospectivos que comparan la evolución de pacientes distribuidos en dos cohortes: en un brazo pacientes tratados según recomendaciones empíricas generales y en el otro pacientes cuyo tratamiento es guiado por los resultados de las pruebas de resistencia al tratamiento antirretroviral. Los resultados de este último tipo de estudios avalan la realización de dichas pruebas para la selección del tratamiento más eficaz en los pacientes previamente tratados en los cuales no se ha obtenido respuesta virológica completa (28-35). En la Tabla 1 se muestran los principales ensayos aleatorizados controlados que han demostrado el impacto a corto plazo de la detección de resistencia a los antirretrovirales (30-36).
TIPOS DE TÉCNICAS PARA LA DETECCIIN DE RESISTENCIAS Existen dos grandes grupos de técnicas para la detección de resistencias del VIH al tratamiento antirretroviral: genotípicas y fenotípicas. Las pruebas genotípicas se basan en el análisis del genoma y por tanto detectan la presencia de mutaciones. En función del principio en que se basen, el número de mutaciones detectables es distinto (14, 19, 37). Las técnicas que utilizan la secuenciación detectan todas las mutaciones presentes en las regiones del genoma del VIH que codifican para la retrotranscriptasa y para la proteasa. Cuando el fundamento de la técnica es la hibridación, sólo se detecta determinado número de mutaciones de significación conocida (38). Las pruebas fenotípicas consisten en un sistema de replicación in vitro que enfrenta al virus con diferentes concentraciones de fármacos antirretrovirales. El grado de inhibición del crecimiento se establece por comparación con una cepa de referencia (39). Pruebas genotípicas comerciales Existen tres pruebas genotípicas comerciales, la secuenciación y dos técnicas basadas en la hibridación (LiPATM y GeneChipTM), cuyo fundamento describiremos brevemente a continuación. Secuenciación El principio general de esta técnica consiste en la obtención de moléculas de DNA cada una un nucleótido más larga que la precedente, y su posterior separación mediante electroforesis en gel de poliacrilamida (PAGE) de elevada resolución (40). Actualmente, aunque el principio básico no ha cambiado, las modernas técnicas de secuenciación derivan del método de Sanger (41), según el cual en lugar de fraccionar el DNA diana se procede a la síntesis de pequeños fragmentos a partir de la hebra molde. Esta síntesis tiene una peculiaridad, que es la incorporación de nucleótidos terminadores de cadena. Estos nucleótidos en realidad son didesoxinucleótidos, es decir, carecen de grupo hidroxilo (OH) en posición 3', que es fundamental para la elongación de la cadena, ya que es precisamente en esta posición donde la polimerasa inserta el siguiente nucleótido durante la síntesis de DNA. La adición de la proporción adecuada de nucleótidos naturales con respecto a estos didesoxinucleótidos dará lugar a la síntesis de fragmentos de diversos tamaños. Para secuenciar el VIH se pueden adoptar dos estrategias: la secuenciación del genoma proviral integrado en los linfocitos, que ya está como DNA, o la realización de una retrotranscripción previa de RNA plasmático (42). Para poder obtener secuencias fiables, un requisito fundamental es la idoneidad del material de partida, tanto en pureza como en cantidad. De cara a garantizar este aspecto se realiza, en primera instancia, una PCR "anidada", que no es otra cosa que dos PCR seguidas, de forma que el amplificado conseguido es mucho más abundante que si se realizase una PCR ordinaria. Tras la obtención de material genético suficiente se procede a la purificación del amplificado para eliminar restos de iniciadores, sales, enzimas, nucleótidos, etc. Actualmente la técnica más empleada es la secuenciación cíclica, de desarrollo más reciente (43). Se basa en aplicar la tecnología de la PCR al método de Sanger anteriormente descrito. Se realizan cuatro reacciones de PCR en paralelo y en cada una de ellas se incluyen en la mezcla de reacción los siguientes reactivos: - DNA a secuenciar. - Iniciadores específicos: regiones de la retrotranscriptasa y la proteasa (gen pol). - Polimerasa. - Desoxinucleótidos (dNTP) naturales (dATP, dCTP, dGTP, dTTP). - Uno de los cuatro nucleótidos terminadores o didesoxinucleótidos (ddNTPs) (ddATP, ddCTP, ddGTP, ddTTP). De esta forma, en cada reacción se van incorporando nucleótidos naturales hasta que de forma aleatoria se incorpora un nucleótido terminador y cesa la síntesis, de manera que se sintetizan hebras de longitud variable. Una condición de reacción importante es que la adición de los ddNTP a la mezcla de reacción sea en proporción no saturante, ya que en caso contrario se incorporarían estos nucleótidos terminadores en todas las posiciones disponibles y se truncaría la síntesis de DNA. Los fragmentos resultantes llevan marcaje fluorescente y en cada una de las cuatro reacciones se usa un fluorocromo distinto, por lo que los fragmentos que terminan en cada uno de los cuatro ddNTP producirán una señal distinta. Una vez obtenidos todos los fragmentos se procede a su separación electroforética, que se realiza de forma automatizada en un secuenciador, y ésta puede ser según la clásica PAGE de alta resolución o mediante electroforesis capilar. Independientemente de la técnica de electroforesis acoplada al secuenciador, los fragmentos se separarán en función de su peso molecular. Las señales se recogen en un procesador electrónico acoplado al secuenciador y éste deduce la secuencia por comparación de los fragmentos solapantes, de forma que esta secuencia en realidad es una secuencia consenso que representa la secuencias de las subpoblaciones virales mayoritarias. Para detectar la presencia de mutaciones que confieren resistencia a los antirretrovirales se extrae el genoma de una muestra del paciente y se procede a su secuenciación. Una vez obtenida la secuencia del VIH de la muestra problema en forma de electroferograma, ésta se compara con la secuencia de una cepa salvaje de VIH mediante un programa informático acoplado al secuenciador (44, 45). LiPATM El fundamento de esta técnica es una hibridación reversa post-PCR (46, 47). El soporte de la hibridación consiste en tiras de nitrocelulosa con sondas de oligonucleótidos específicos inmovilizadas en líneas paralelas. Estas sondas son complementarias a la secuencia de mutaciones conocidas que confieren resistencia a los fármacos antirretrovirales (43, 48). La amplificación de la muestra se realiza con iniciadores biotinilados que permitirán el revelado de la reacción. Tras la hibridación se añade un conjugado compuesto por estreptavidina-fosfatasa alcalina, que se unirá a cualquier híbrido formado sobre la tira. Al añadir el sustrato de la enzima se producirá un precipitado marrón-púrpura en las posiciones correspondientes. GeneChipTM A pesar de que el fundamento de esta técnica es una hibridación, el resultado final es la secuenciación del material amplificado a partir de la muestra. El soporte de hibridación son "chips" de sílice que contienen un "biblioteca combinatoria" de sondas, que consiste en una gran cantidad de oligonucleótidos solapantes (409.000/1,28 cm2). La hibridación en microarrays va a permitir identificar el nucleótido presente en cada posición de la secuencia a analizar (49, 50). Puesto que los productos de RT-PCR están marcados, la detección de fluorescencia tras la hibridación mediante barrido de láser e integración de las señales obtenidas con un programa informático permitirá deducir la secuencia. Una ventaja de esta técnica es que permite el análisis simultáneo de un elevado número de muestras. En la Tabla 2 se muestran las principales ventajas e inconvenientes de las pruebas genotípicas comerciales.
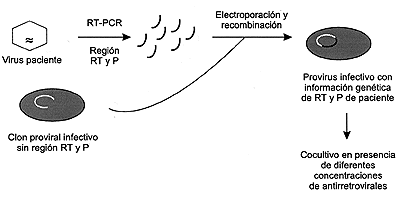
Pruebas fenotípicas comerciales Dado que las pruebas genotípicas son una medida indirecta, pues la presencia de mutaciones no siempre implica su expresión, las pruebas fenotípicas deberían ser el primer escalón natural en la detección de resistencias (51, 52). Clásicamente estas técnicas consistían en el cocultivo de linfocitos del paciente con líneas celulares linfoides, como MT2 y MT4, en presencia de fármacos antirretrovirales (53). Transcurridas unas semanas se medía la replicación viral por la producción de Ag p24 o la actividad retrotranscriptasa. Estas técnicas tienen varias limitaciones e inconvenientes, como son la laboriosidad, el alto coste, la infraestructura necesaria, el largo tiempo requerido y la falta de estandarización. Todo esto ha sido solventado en parte por las técnicas de virus recombinantes (RVA) (54), cuyo fundamento se muestra en la Fig. 1.
Figura 1. Fundamento de las técnicas de virus recombinantes. Las técnicas de virus recombinantes determinan la concentración que inhibe el 50% de la infectividad (IC50), y la comparan con la IC50 de una cepa de referencia o de un aislamiento anterior de un mismo paciente. Existen dos técnicas comerciales (55, 56) que se diferencian principalmente en los siguientes aspectos: - Sistema de producción del virus recombinante. - Mecanismo de detección de la replicación viral. - Valor umbral de carga viral requerido. - Tiempo de realización. - Cepa de referencia. - Punto de corte. En la Tabla 3 se muestran las características diferenciales más destacables de las dos técnicas fenotípicas comercializadas.
Entre las principales ventajas que presentan las pruebas fenotípicas de resistencia a los fármacos antirretrovirales se encuentran su capacidad para medir directamente la sensibilidad al fármaco en cuestión, que aportan información sobre resistencias cruzadas, que se pueden aplicar a cualquier molécula y que son relativamente fáciles de interpretar (57, 58), tal y como se refleja en la Tabla 4, donde también se especifican sus inconvenientes más destacados.
La Tabla 5 resume los principales métodos de detección de resistencias a los antirretrovirales, así como su nombre comercial y compañía distribuidora.
En la Tabla 6 se recogen las principales características de las técnicas de detección de resistencias que están actualmente disponibles. Tal y como se puede observar, la sensibilidad interpretada como la proporción exigible a una subpoblación viral para ser detectable oscila entre un 1% y un 25%, y la complejidad técnica es, salvo con LiPA, alta. El umbral mínimo de carga viral exigible para su realización se sitúa en general en torno a las 1000 copias de ARN/ml, y el tiempo necesario para obtener el resultado oscila entre dos o tres días y tres semanas (59). Limitaciones de las pruebas de detección de resistencias Una precaución importante a tener en cuenta a la hora de tomar las muestras para estas determinaciones es asegurar que en ese momento el paciente continúa con el tratamiento que supuestamente ha fracasado (60). Algunos autores recomiendan que el plazo máximo transcurrido entre el cese del tratamiento y la detección de resistencias no supere los 15 días (61), pues el rápido recambio de la población viral conduciría a un predominio de subpoblaciones salvajes que daría lugar a falsos negativos en la determinación de resistencias (62). Dentro de las limitaciones que en general presentan estas técnicas, independientemente del principio en que se basen, está la proporción mínima que debe representar una subpoblación determinada respecto al global de la muestra (63). A pesar de que varía en función de la sensibilidad de la técnica, se puede decir que se necesita una proporción de un 20% para que esa subpoblación esté representada en el conjunto, y por tanto sea detectable. Además, se requiere un umbral de carga viral mínimo para garantizar la obtención de resultados fiables, que generalmente se establece en 1000 copias de ARN/ml, aunque algunas técnicas aseguran que se podría rebajar este umbral a 500 copias de ARN/ml (64). Como ya hemos expuesto, ambos tipos de técnicas de detección de resistencias poseen ventajas y desventajas, pero en principio las genotípicas parecen más rápidas, asequibles y baratas, aunque utilizar sus resultados para predecir la respuesta a los fármacos puede resultar un poco prematuro por varios factores: presencia de mezclas de subpoblaciones no detectadas, conocimiento incompleto de las mutaciones que confieren resistencia, interacción de diferentes mutaciones, resistencias cruzadas y respuesta variable a los fármacos en función de los pacientes (65, 66). Por todo ello, es necesario definir mejor el valor predictivo de este tipo de técnicas. Por el contrario, las fenotípicas son una medida directa de la resistencia, pero su interpretación también es difícil. Poseen la ventaja de determinar la influencia de mutaciones múltiples en el genoma, a diferencia de las técnicas genotípicas, pero también por ello el papel individual de una mutación determinada puede quedar enmascarado (67). Otro inconveniente de estas últimas técnicas es que para que llegue a detectarse fenotípicamente una resistencia es necesario que una proporción suficientemente grande de la muestra posea determinadas mutaciones. RECOMENDACIONES PARA LA DETECCIÓN DE RESISTENCIAS A LOS ANTIRRETROVIRALES Desde diferentes organismos, tanto nacionales como internacionales, se han emitido recomendaciones para la detección de resistencias a los antirretrovirales (68). Entre las guías internacionales destacan cuatro como ineludibles: las emitidas por el DHHS Panel on Clinical Practices for the Treatment of HIV Infections (69), las de la British HIV Association (70) y las publicadas por Hirsch y cols. (71) y por Schinazi y cols. (72). En el ámbito nacional, la Secretaría del Plan Nacional sobre el Sida (SPNS) ha elaborado dos documentos al respecto, el primero de ellos en 1997 (73) y el último en 2000 (74). También han emitido sus recomendaciones el Panel Español de Resistencia a los Antirretrovirales (75) y el grupo GESIDA (76, 77) junto con la SPNS. No es nuestro propósito revisar de manera exhaustiva las referidas recomendaciones y remitimos al lector interesado a los documentos originales. La SPNS centra actualmente su interés en cuatro aspectos (74, 76, 77). En primer término, el proyecto de realización de una base de datos con el fin de aglutinar los resultados de las pruebas de resistencia realizadas por los laboratorios españoles, de forma que su diseño y gestión se lleven a cabo en colaboración con el Centro de Biología Molecular Severo Ochoa. En segundo lugar trabaja en un Programa de Control de Calidad que ha sido ofertado a los laboratorios que realizan estas técnicas, y cuya logística es similar a la se que viene realizando desde hace años en la cuantificación de la carga viral. Este programa será coordinado por el Instituto de Salud Carlos III y la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC). En tercer lugar, persigue la optimización del uso de estas técnicas fomentando, por una parte, la docencia en este ámbito a través de cursos, seminarios y jornadas, y por otra parte el entrenamiento del personal responsable de su ejecución. En último término, impulsa la consolidación de agencias de investigación con esta línea prioritaria, de forma que se logre una utilización eficiente y se estudien indicaciones futuras de estas técnicas, como son la epidemiología de las resistencias primarias y el empleo de las pruebas fenotípicas. INCORPORACIÓN DE LAS PRUEBAS DE RESISTENCIAS A LA ASISTENCIA La incorporación selectiva de las técnicas genotípicas a la práctica asistencial tiene interés, primero, por el beneficio individual de cada paciente, ya que su filosofía es optimizar e individualizar el tratamiento (77). En sentido colectivo, esta incorporación también tiene utilidad ya que permite conocer la "epidemiología" de las cepas de VIH resistentes, incidir en la mejor utilización del arsenal terapéutico disponible e intentar disminuir el riesgo de aparición de resistencias potencialmente transmisibles (78). La SPNS, respecto a la implantación de estas técnicas, tiene como objetivo asegurar su homogeneidad y validez, para lo que señala cuatro situaciones en que está indicada la detección de resistencias genotípicas (76, 77): 1) Pacientes adultos de quienes se supone que están realizando correctamente el tratamiento y se plantea llevar a cabo un cambio de antirretrovirales ante un fracaso terapéutico. La detección de resistencias se debe considerar en un primer fracaso y se debe recomendar tras un segundo o posterior fracaso terapéutico. 2) En los casos con primoinfección sintomática, especialmente si se va a iniciar un tratamiento antirretroviral. 3) En las mujeres embarazadas, muy especialmente si la supresión de la replicación viral por el tratamiento no es suficiente, con el fin de conseguir la pauta terapéutica óptima y la máxima reducción de la transmisión vertical del VIH. 4) En los niños con nuevo diagnóstico de infección por VIH y en aquellos que ya están recibiendo tratamiento antirretroviral si presentasen criterios de fracaso terapéutico. Uno de los aspectos importantes en relación a la incorporación a la práctica asistencial de estas técnicas es la definición de "áreas preferentes de estudio" que contribuyan a mejorar el conocimiento en este campo, de cara a dilucidar temas que aún no están del todo claros. En este sentido, han de realizarse estudios retrospectivos que correlacionen el genotipo o el fenotipo, o ambos, con la respuesta virológica (79). Son también necesarios estudios prospectivos sobre el valor que tiene aportar información del genotipo o fenotipo, así como estudios prospectivos de diseño de terapias de rescate basadas en resultados de genotipo o fenotipo (80, 81). En cuanto a la estimación de la presencia de mutaciones preexistentes (resistencias primarias), han de realizarse estudios epidemiológicos en pacientes naïve con infección aguda o seroconversión reciente (58, 82). Desde un enfoque que contemple la realización de estas pruebas en el laboratorio de diagnóstico microbiológico resulta determinante adoptar una serie de recomendaciones, que bajo el epígrafe de "buena práctica" reflejamos en la Tabla 7. En primer lugar, es importante que el laboratorio que oferta la técnica elabore unas normas de recogida, envío y transporte de muestras, cuya distribución es preceptiva entre los posibles demandantes de la técnica (83). En estrecha relación con la actividad precedente se encuentra la elaboración y el diseño de un modelo específico de volante, que en nuestra experiencia debe ser incluido entre los que habitualmente se emiten desde el Servicio de Microbiología. Es deseable una discusión interdisciplinaria de los parámetros en él incluidos y referidos al menos a cuatro tipos de variables: identificativas, epidemiológicas, clínicas y de la historia de la terapia seguida (84). Resulta de inestimable valor disponer de una muestra inicial de plasma de cara a poder delimitar con precisión, en los casos en que no se hubiera hecho con anterioridad, la existencia de marcadores de infección por VIH (85). Obviamente, es preciso disponer de una infraestructura adecuada que cumpla con los requisitos exigibles para el desarrollo de este tipo de técnicas encuadradas dentro del ámbito de lo que podríamos denominar "diagnóstico microbiológico molecular" (86). Una ayuda adicional al manejo y gestión tanto de la información recibida como de la generada es la proporcionada por la disponibilidad de una base de datos específicamente diseñada al efecto. En este sentido, las actuales redes de Intranet y las que sirven al laboratorio de microbiología deberían hacer el esfuerzo de incorporar elementos para una gestión ágil e integrada de este tipo de información (59, 61). Finalmente, parece oportuno abordar el difícil reto, no siempre resuelto, de emitir un informe en términos inteligibles que facilite al clínico que prescribe tratamientos antirretrovirales la toma de decisiones eficientes (78, 81, 83).
Tabla 7. Aspectos de "buena práctica" en el laboratorio que deben acompañar a la implantación de las pruebas de resistencia del VIH a los fármacos antirretrovirales. CUESTIONES PENDIENTES A pesar de la intensa actividad que se
está desarrollando, es importante asumir que hay una serie de problemas
de muy diversa índole que, lejos de estar definitivamente resueltos,
requieren una mantenida atención. Para el profesional del laboratorio existen,
en primer lugar, algunas cuestiones inquietantes relativas a los aspectos
técnicos, entre las que se incluyen la baja rentabilidad de la
amplificación (14, 87), la deficiente estandarización de
determinados procedimientos (43, 59, 64, 88) y la escasa implantación
de los programas de control de calidad (28, 89). En estrecha vinculación
con estos aspectos se sitúa, a continuación, el reto de
la interpretación de los hallazgos del laboratorio, cuya expresión
más paradigmática la constituye el hecho de valorar las
relaciones entre mutaciones y aparición de resistencias cruzadas
(81, 90, 91). Corresponde al profesional clínico validar en la
práctica real la información generada por el microbiólogo
(83, 84) e impulsar el desarrollo de ensayos que contribuyan a esclarecer
el verdadero peso de esta información (92, 93). En nuestra opinión existen al
menos seis aspectos relativos a la detección de resistencias del
VIH cuyo esclarecimiento no debe ser demorado (Tabla 8). Es preciso establecer
cuáles son los beneficios constatables a largo plazo en términos
de evolución virológica. A continuación, parece pertinente
delimitar en qué situaciones debe optarse por una prueba genotípica
o fenotípica. En tercer lugar, convendría describir las
características intrínsecas de sensibilidad de las diversas
técnicas para detectar subpoblaciones virales minoritarias. Sería
oportuno también disponer de un método de evaluación
específico para delimitar las resistencias atribuibles a cada fármaco
en el contexto de regímenes combinados. Otro punto pendiente de
resolución es determinar la cadencia de las determinaciones de
resistencias. En última instancia, parece inexcusable impulsar
estudios de coste-efectividad en la aplicación asistencial de las
pruebas de resistencia. Uno de los aspectos relativos a la determinación
de resistencias, que por su trascendencia requiere un apartado específico,
es la interpretación de los resultados de estas técnicas
(19, 78, 81, 83, 94). No existe una pauta acerca de cuál sea la
mejor interpretación de las pruebas. Entre las posibilidades disponibles
se encuentran la interpretación interdisciplinaria, que implica
la participación de microbiólogos, clínicos y farmacólogos
(95), y los sistemas basados en reglas generadas por la evidencia científica
y la realización del denominado "fenotipo virtual" (58, 59, 96).
En la práctica se usan estos tres tipos de interpretaciones, que
probablemente aporten sus beneficios, pero se necesitan estudios que las
comparen con el fin de hallar el mejor método de interpretación. Tanto las técnicas genotípicas
como las fenotípicas presentan problemas a la hora de interpretar
sus resultados. Las pruebas genotípicas aportan información
sobre "posibles" resistencias, en el sentido de que no se puede garantizar
la expresión de las mutaciones detectadas, y por ello requieren
guías de interpretación basadas en la mejor evidencia científica
disponible (68, 78, 81, 84, 97, 98). Además, estas técnicas
poseen mayor valor para detectar resistencias, considerando las mutaciones
presentes, que para predecir sensibilidad, para lo cual sería necesario
descartar todas las posibles causas de resistencia. Un hecho que reviste
especial dificultad es la interpretación de patrones de varias
mutaciones (99). Por su parte, las técnicas fenotípicas
constituyen una medida de la replicación viral en presencia de
fármacos antirretrovirales, y ésta responde a diversos factores,
unos conocidos y otros no (18, 19, 100). Además, en este sentido,
los puntos de corte para determinar la resistencia no están ni
bien definidos ni clínicamente validados. Otro factor que dificulta
la interpretación de los resultados es el desconocimiento de los
niveles mínimos requeridos in vivo para lograr la supresión
de la replicación viral (101), lo cual aún resulta más
complejo en el caso de los inhibidores de la retrotranscriptasa análogos
de nucleósido, ya que éstos se activan por trifosforilación
en la célula diana, de forma que la concentración activa
de fármaco ha de medirse en el interior de la célula, con
la mayor dificultad que esto entraña (102, 103). Una consideración que conviene
tener presente es el hecho de que existen distintas causas a las que achacar
el fracaso virológico (76, 104, 105): al paciente, al fármaco
y al VIH. La principal causa de fracaso atribuible al paciente es el mal
cumplimiento de la pauta prescrita (106, 107). De los aspectos relativos
al fármaco destacamos, por un lado, parámetros farmacocinéticos,
y por otro factores farmacológicos. Dentro de los primeros, el
más directamente asociado al fracaso es una absorción insuficiente
del fármaco, que condicionará concentraciones plasmáticas
subterapéuticas (104, 107). Con respecto a los factores farmacológicos,
podemos encontrar asociadas al fracaso virológico circunstancias
como una insuficiente potencia antiviral intrínseca de la combinación
de fármacos, interacciones de antirretrovirales y otros fármacos,
y una activación farmacológica inadecuada (108, 109). Por
último, el fracaso puede estar condicionado por causas atribuibles
al propio VIH, como son la replicación en compartimientos "santuario"
y el desarrollo de resistencias (78, 110, 111). Por tanto, a pesar de
la importancia que posee la determinación de resistencias al tratamiento
antirretroviral, no debemos olvidar que existen otras causas de fracaso
que también han de ser exploradas. Para finalizar nuestra reflexión,
queremos hacer hincapié en que nos encontramos en un campo en constante
evolución, que exige una continua puesta al día debido al
desarrollo de nuevos fármacos y nuevas dianas terapéuticas,
la descripción de nuevas mutaciones asociadas a resistencia y la
acumulación y jerarquización de evidencia científica.
Existen bases de datos de mutaciones que confieren resistencia al VIH
de alimentación continua y fácil consulta a través
de varias páginas web de Internet, que en este sentido nos
facilitan enormemente la actualización de conocimientos. Asimismo,
las técnicas de detección de resistencia se verán
obligadas a asumir este dinamismo, adaptándose a los datos que
se vayan generando. Por tanto, cada vez tendremos que tener en cuenta
más factores a la hora de interpretar los resultados de la detección
de resistencias del VIH a los fármacos antirretrovirales. Por último, esperamos que en la
medida en que avance el conocimiento seamos capaces de superar las dificultades
técnicas, de interpretación, de interdisciplinariedad y
de implementación asistencial que actualmente existen en este campo. Parte del presente trabajo se expuso en
la reunión que sobre resistencias del VIH se desarrolló
bajo los auspicios de la Sociedad Española de Quimioterapia el
15 de diciembre de 2000, en el Colegio Oficial de Médicos de Madrid. Correspondencia: José M.
Eiros Bouza, Área de Microbiología Sexta Planta, Facultad
de Medicina, Avda. Ramón y Cajal 7, 47005 Valladolid. BIBLIOGRAFÍA 1. Balzarini, J., Pelemans, H., De Clerq,
E., Karlsson, A., Kleim, J.P. Reverse transcriptase fidelity and HIV-1
variation. Science 1997; 275: 229-230. 2. Turner, B.G., Summers, M.F. Structural
biology of HIV. J Mol Biol 1999; 285: 1-32. 3. Katz, R.A., Skalka, A.M. The retroviral
enzymes. Annu Rev Biochem 1994; 63: 133-173. 4. Perelson, A.S., Neumann, A.U., Markowitz,
M., Leonard, J.M., Ho, D.D. HIV-1 dynamics in vivo: Virion clearance
rate, infected cell life-span, and viral generation time. Science
1996; 271: 1582-1586. 5. Coffin, J.M. HIV population dynamics
in vivo: Implications for genetic variation, pathogenesis and therapy.
Science 1995; 267: 483-489. 6. Domingo, E., Holland, J.J. RNA
virus mutations and fitness for survival. Annu Rev Microbiol 1997;
51: 151-178. 7. Drake, J.M. Rates of spontaneous
mutations among RNA viruses. Proc Natl Acad Sci USA 1993; 90: 4171-4175. 8. Wei, X., Ghosh, S.K., Taylor, M.E.
y cols. Viral dynamics in HIV-1 infection. Nature 1995; 373: 117-122. 9. Domingo, E., Menéndez-Arias,
L., Quiñones-Mateu, M.E. y cols. Viral quasispecies and the
problem of vaccine-escape and drug-resistant mutants. Prog Drug Res
1997; 48: 99-128. 10. Soriano, V., Domingo, E. Importancia
clínica de la variedad genética del VIH. Med Clin (Barc)
1996; 107: 460-463. 11. Telenti, A., Muñoz, M., Bleiber,
G. y cols. Heterogeneity in the response to antiretroviral therapy.
AIDS Rev 1999; 1: 147-155. 12. Descamps, D., Flandre, P., Calvez,
V. y cols. Mechanisms of virologic failure in previously untreated
HIV-infected patients from a trial of induction-maintenance therapy.
JAMA 2000; 283: 205-211. 13. Wainberg, M.A., Friedland, G. Public
health implications of antiretroviral therapy and HIV drug resistance.
JAMA 1998; 279: 1977-1983. 14. Briones C. Resistencias del VIH
a los fármacos antirretrovirales. En: Soriano, V., González-Lahoz,
J. (Eds.). Curso de biología molecular para clínicos. Permanyer,
Barcelona 2000; 87-100. 15. Alcamí, J., Delgado, R., Menéndez-Arias,
L. Bases biológicas de la generación de resistencias
a fármacos antirretrovirales. Enferm Infecc Microbiol Clin
2001; 19 (monogr.): 3-13. 16. Condra, J.H. Resisting resistance.
Ann Intern Med 1998; 128: 951-954. 17. Pomerantz, R.J. Primary HIV-1 resistance.
A new phase in the epidemic? JAMA 1999; 282: 1177-1179. 18. Soriano Vázquez, V., Rodríguez-Rosado,
Martínez-Echevarría, R. Introducción de las pruebas
de detección de resistencias a los antirretrovirales en la práctica
clínica. Rev Clin Esp 1999; 199: 179-183. 19. Rodríguez-Rosado, R., Briones,
C., Soriano, V. Introduction of HIV drug resistance testing in clinical
practice. AIDS 1999; 13: 1007-1014. 20. Richman, D.D., Grimes, J.M., Lagakos,
S.W. Effect of stage of disease and drug dose on zidovudine susceptibilities
of isolates of human immunodeficiency virus. J Acquir Immune Defic
Syndr 1990; 3: 743-746. 21. St. Clair, M.H., Martin, J.L., Tudor-Williams,
G. y cols. Resistance to ddI and sensitivity to AZT induced by a mutation
in HIV-1 reverse transcriptase. Science 1991; 253: 1557-1559. 22. Kellam, P., Boucher, C.A., Larder,
B.A. Fifth mutation in human immunodeficiency virus type 1 reverse
transcriptase contributes to the development of high level resistance
to zidovudine. Proc Natl Acad Sci USA 1992; 89: 1934-1938. 23. Zhang, D., Caliendo, A.M., Eron, J.J.
Resistance to 2',3'-dideoxycytidine conferred by a mutation in codon
65 of the human immunodeficiency virus type 1 reverse transcriptase.
Antimicrob Agents Chemother 1994; 38: 282-287. 24. Lacey, S.F., Larder, B.A. Novel
mutation (V75T) in human immunodeficiency virus type 1 reverse transcriptase
confers resistance to 2',3'- didehydro-2',3'-dideoxy-thymidine in cell
culture. Antimicrob Agents Chemother 1994; 38: 1428-1432. 25. Johson, V.A. Nucleoside reverse
transcriptase inhibitors and resistance of human immunodeficiency virus
type 1. J Infect Dis 1995; 171 (Suppl. 2): S140-S149. 26. Eron, J.J., Benoit, S.L., Jemsek,
J. y cols. Treatment with lamivudine, zidovudine or both in HIV positive
patients with 200 to 500 CD4+ cells per cubic millimeter. North
American HIV Working Party. N Engl J Med 1995; 333: 1662-1669. 27. Huang, H., Chopra, R., Verdine, G.L.,
Harrison, S.C. Structure of a covalently trapped catalytic complex
of HIV-1 reverse transcriptase: Implications for drug resistance.
Science 1998; 282: 1669-1675. 28. Schuurman, R., Brambilla, D., De Groot,
T., Boucher, C. Second worldwide evaluation of HIV-1 drug resistance
genotyping quality using the ENVA-2 panel. Antiviral Therapy 1999;
4 (Suppl. 4): S41. 29. Cooper, D.A., Emery, S. Therapeutic
strategies for HIV infection. Time to think hard. N Engl J Med 1998;
339: 1319-1321. 30. Reijers, M.H.E., Wevering, G.J., Jurriaans,
S. y cols. Maintenance therapy after quadruple induction therapy in
HIV infected individuals: Amsterdam Duration of Antiretroviral Medication
(ADAM) Study. Lancet 1998; 352: 185-190. 31. Durant, J., Clevenbergh, P., Halfon,
P. y cols. Drug-resistance genotyping in HIV-1 therapy: The VIRADAPT
randomised trial. Lancet 1999; 353: 2195-2200. 32. Baxter, J.D., Mayers, D.L., Wenworth,
D.N. y cols. Final results of CPCRA 046: A pilot study of antiretroviral
management based on plasma genotypic antirretroviral resistance testing
(GART) in patients failing antiretroviral therapy. Antiviral Ther
1999; 4 (Suppl. 1): 43, Abstr. 61. 33. Cohen, C., Hunt, S., Sension, M. y
cols. Phenotypic resistance testing significantly improves response
to therapy (Tx). 7th Conference on Retroviruses and Opportunistic
Infections, San Francisco, CA 2000; Abstr. 237. 34. Brodine, S., Shaffer, R., Starkey,
M. y cols. Drug resistance patterns, genetic subtypes, clinical features,
and risk factors in military personnel with HIV-1 seroconversion.
Ann Intern Med 1999; 131: 502-506. 35. Meynard, J.L., Vray, M., Morand-Joubert,
J. y cols. Impact of treatment guided by phenotypic or genotypic resistance
tests on the response to antiretroviral therapy (ART): A randomised trial
(NARVAL, ANRS 088). 40th Interscience Conference on Antimicrobial
Agents and Chemotherapy, Toronto, Canada 2000; Abstr. 698. 36. Tural, C., Ruiz, L., Holtzer y cols.
The potencial role of resistance decision support software with or
without expert advice in a trial of HIV genotyping versus standard of
care - The Havanna trial. 40th Interscience Conference on Antimicrobial
Agents and Chemotherapy, Toronto, Canada 2000; Abstr. L-10. 37. Kuritzes, D.R. Drug resistance
testing: Time to be used in clinical practice? AIDS Rev 1999; 1: 45-50. 38. Eastman, P.S., Urdea, M., Besemer,
D., Stempein, M., Kolberg, J. Comparison of selective polymerase chain
reaction primers and differential probe hybridation of polymerase chain
reaction products for determination of relative amounts of codon 215 mutant
and wild-type HIV-1 populations. J Acquir Immune Defic Syndr Hum Retrovirol
1993; 9: 264-269. 39. De Jong, M.D., Schuurman, R., Lange,
J.M.A., Boucher, C.A.B. Replication of pre-existing resistant HIV-1
subpopulation in vivo after introduction of a strong selective drug presure.
Antiviral Therapy 1996; 1: 33-41. 40. Innis, M., Myambo, K., Gelfand, D.
y cols. DNA sequencing with Thermus acquaticus DNA polymerase and direct
sequencing of polymerase chain reaction-amplified DNA. Proc Natl Acad
Sci USA 1988; 85: 9436-9440. 41. Sanger, F., Nicklens, S., Coulson,
A. DNA sequencing with chain-termination inhibitors. Proc Natl
Acad Sci USA 1977; 74: 5463-5467. 42. Briones, C. Secuenciación.
En: Soriano, V., González-Lahoz, J. (Eds.). Curso de biología
molecular para clínicos. Permanyer, Barcelona 2000; 43-50. 43. Ruiz, L., Clotet, B. Test de resistencias
y sus limitaciones técnicas. En: Grupo E. Entheos (Ed.). Guía
práctica para el manejo de las resistencias al VIH. Gráficas
Marte, Madrid 1999; 29-55. 44. National Center for Biotechnology
Information. National Library of Medicine. National Institutes of Health.
GenBank: http://www.ncbi. nlm.nih.gov/ 45. Antiviral Drug Resistance On Line.
http://www.viral-resistance.com 46. Stuyver, L., Wyseur, A., Rombout,
A. y cols. Line probe assay for rapid detection of drug-selected mutations
in the human immunodeficiency virus type 1 reverse transcriptase gene.
Antimicrob Agents Chemother 1997; 41: 283-291. 47. Schmit, J.C., Ruiz, L., Stuyver, L.
y cols. Comparison of the LiPA HIV 1 RT test, selective PCR and direct
solid phase sequencing for the detection of HIV-1 drug resistance mutations.
J Virol Methods 1998; 73: 77-82. 48. Jacobsen, H., Yasargil, K., Winslow,
D.L. y cols. Characterization of human immunodeficiency virus type
1 mutants with decreased sensitivity to proteinase inhibitor Ro-31-8959.
Virology 1995; 206: 527-534. 49. Kuzal, M.J., Shah, N., Shen, N. y
cols. Extensive polymorphisms observed in HIV-1 clade B protease gene
using high-density oligonucleotide arrays. Nat Med 1996; 2: 753-759. 50. Gunthard, H.F., Wong, J.K., Ignacio,
C.C., Havlir, D.V., Richman, D.D. Comparative performance of high-density
oligonucleotide sequencing and dideoxynucleotide sequencing of HIV type
1 pol from clinical samples. AIDS Res Human Retroviruses 1998; 14:
869-876. 51. Brun-Vezinet, F., Ingrand, D., Deforges,
L. y cols. HIV-1 sensitivity to zidovudine: A consensus culture technique
validated by genotypic analysis of the reverse transcriptase. J Virol
Methods 1992; 37: 177-188. 52. Petropoulus, C.J., Parkin, N.T., Limoli,
K.L. A novel phenotypic drug susceptibility assay for human immunodeficiency
virus type 1. Antimicrob Agents Chemother 2000; 44: 920-928. 53. Japour, A.J., Mayers, D.L., Johnson,
V.A. y cols. Standarized peripheral blood mononuclear cell culture
assay for determination of drug susceptibilities of clinical human immunodeficiency
virus type 1 isolates. The RV-43 Study Group, The AIDS Clinical Trials
Group Virology Committee Resistance Working Group. Antimicrob Agents
Chemother 1993; 37: 1095-1101. 54. Kellam, P., Larder, B.A. Recombinant
virus assay: A rapid, phenotypic assay for assesment of drug susceptibility
of human immunodeficiency virus type 1 isolates. Antimicrob Agents
Chemother 1996; 38: 23-30. 55. Hertogs, K., de Bethume, M.P., Miller,
V. y cols. A rapid method for simultaneous detection of phenotypic
resistance to inhibitors of protease and reverse transcriptase in recombinant
human immunodeficiency virus type 1 isolates from patients treated with
antiretroviral drugs. Antimicrob Agents Chemother 1998; 42: 269-276. 56. Ruiz, L., Martínez-Picado,
J., Clotet, B. Resistencia del VIH a los antirretrovirales. En:
Gatell Artigas, J.M., Clotet Salas, B., Podzamczer Palter, D., Miró
Meda, J.M., Mallolas Masferrer, J. (Eds.). Guía práctica
del SIDA. Clínica, diagnóstico y tratamiento, 6ª ed.
Masson, Barcelona 2000; 413-426. 57. Bacheler, L., Jeffrey, S., Hanna,
G. y cols. Genotypic correlates of phenotypic resistance to efavirenz
in virus isolates from patients failing nonnucleoside reverse transcriptase
inhibitor therapy. J Virol 2001; 75: 4999-5008. 58. Boden, D., Hurley, A., Zhan, L. y
cols. HIV-1 drug resistance in newly infected individuals. JAMA
1999; 282: 1135-1141. 59. Erice, A., Pérez, J.L., Pumarola,
T. Análisis comparativo de los métodos de detección
de resistencias. Enferm Infecc Microbiol Clin 2001; 19 (monogr.):
14-21. 60. Martínez-Picado, J., Clotet,
B. Mutaciones de resistencia seleccionadas durante tratamiento antirretroviral.
En: Grupo E. Entheos. Guía práctica para el manejo clínico
de las resistencias al VIH. Gráficas Marte, Madrid 1999; 57-133. 61. Moreno, S. Los estudios de resistencias
son útiles para guiar el tratamiento antirretroviral. Enferm
Infecc Micrbiol Clin 2001; 19 (monogr.): 42-44. 62. Eigem, M. On the nature of viral
quasispecies. Trends Microbiol 1996; 4: 212-214. 63. Telenti, A. Determinación
de susceptibilidad al VIH: Una posición (en contra) matizada respecto
a la tecnología compleja. Enferm Infecc Microbiol Clin 2001;
19 (monogr.): 45-46. 64. Ruiz, L., Martínez-Picado,
J., de Mendoza, C., Soriano, V., Clotet, B. Test de resistencias. Ventajas
y limitaciones. En: Clotet, B., Menéndez-Arias, L., Ruiz, L.
y cols. (Eds.). Guía para el manejo de las resistencias al VIH
y de la farmacocinética de los antirretrovirales, 1ª ed. TAISA,
Barcelona 2000; 9-20. 65. Flexner, C. HIV genotype and phenotype-arresting
resistance? JAMA 2000; 283: 1177-1179. 66. Perrin, L., Telenti, A. HIV treatment
failure: Testing for HIV resistance in clinical practice. Science
1998; 280: 1871-1873. 67. Menéndez-Arias, L., Domingo,
E. Cambios de aminoácido asociados a resistencia a inhibidores
de la retrotranscriptasa y de la proteasa del VIH-1. En: Clotet, B.,
Menéndez-Arias, L., Ruiz, L. y cols. (Eds.). Guía para el
manejo de las resistencias al VIH y de la farmacocinética de los
antirretrovirales, 1ª ed. TAISA, Barcelona 2000; 30-55. 68. Antela, A., Iribaren, J.A., Clotet,
B., Guerra, L. Documentos de consenso y guías clínicas
sobre resistencias a antirretrovirales. Enferm Infecc Microbiol Clin
2001; 19 (monogr.): 47-52. 69. DHHS Panel on Clinical Practices for
the Treatment of HIV Infections. Guidelines for the use of antiretroviral
agents in HIV infected adults and adolescents. Washington 2000; http://www.hivatis.org 70. Pozniac, A., Gazzard, B., Churchill,
D. on behalf of the BHIVA Executive Committee. British HIV Association
(BHIVA) guidelines for the treatment of HIV-infected adults with antiretroviral
therapy. London 2000; http://www.aidsmap.com 71. Hirsch, M., Brun-Vezinet, F., D'Aquila,
R. y cols. Antirretroviral drug resistance testing in adult HIV-1 infection.
JAMA 2000; 283: 2147-2426. 72. Schinazi, R.F., Larder, B., Mellors,
J.W. Mutations in retroviral genes associated with drug resistance:
2000-2001 update. Internat Antiviral News 2000; 8: 65-91. 73. Secretaría del Plan Nacional
sobre el Sida (SPNS). Las resistencias a los fármacos antirretrovíricos:
Utilización de los test de resistencias en la práctica asistencial.
Madrid 1997; http://www.msc.es/sida/asis tencia/resistencias.htm 74. Secretaría del Plan Nacional
sobre el Sida (SPNS). Las resistencias a los fármacos antirretrovirales:
Utilización de los test en la práctica asistencial. Madrid
2000; http://www.msc.es/sida/asistencia/resisten cias.htm 75. Ledesma, E., Soriano, V. on behalf
of the Spanish Drug Resistance Panel. Spanish consensus on drug resistance
testing in clinical practice. AIDS 1999; 13: 1998-2001. 76. Miró, J., Antela, A., Arrizabalaga,
J. y cols. Recomendaciones de GESIDA/Plan Nacional sobre el Sida respecto
al tratamiento antirretrovírico en pacientes adultos infectados
por el virus de la inmunodeficiencia humana en el año 2000 (I).
Enferm Infecc Microbiol Clin 2000; 18: 329-351. 77. Miró, J., Antela, A., Arrizabalaga,
J. y cols. Recomendaciones de GESIDA/Plan Nacional sobre el Sida respecto
al tratamiento antirretrovírico en pacientes adultos infectados
por el virus de la inmunodeficiencia humana en el año 2000 (II).
Enferm Infecc Microbiol Clin 2000; 18: 396-412. 78. Alcamí, J., Rubio, R. Pruebas
de resistencias a antirretrovirales, un elemento necesario en el seguimiento
del paciente con infección por VIH. Enferm Infecc Microbiol
Clin 2001; 19 (monogr.): 1-2. 79. Lorenzi, P., Opravil, M., Hirschel,
B. y cols. Impact of drug resistance mutations on virologic response
to therapy. AIDS 1999; 13: F17-F21. 80. Zolopa, A.R., Shafer, R.W., Warford,
A. y cols. HIV-1 genotipic resistance patterns predict response to
saquinavir-ritonavir therapy in patients in whom previous protease inhibitor
therapy had failed. Ann Intern Med 1999; 131: 813-822. 81. Vandamme, A.M. Algoritmo para el
uso clínico de las resistencias genotípicas y fenotípicas.
En: Clotet, B., Menéndez-Arias, L., Ruiz, L. y cols. (Eds.). Guía
para el manejo de las resistencias al VIH y de la farmacocinética
de los antirretrovirales, 1ª ed. TAISA, Barcelona 2000; 85-89. 82. Kindelán, J.M., Giménez-Domènech,
Vidal-Verdú, E., Madueño, J. y Grupo Andaluz para el Estudio
de las Enfermedades Infecciosas (GAEI). Prevalencia de mutaciones primarias
en el virus de la inmunodeficiencia humana que confieren resistencia a
análogos de nucleósidos en pacientes de Andalucía
no tratados previamente. Med Clin (Barc) 2000; 115: 423-425. 83. Tural, C., Sirera, G., Romeu, J.,
Ruiz, L., Podzamczer, D., Clotet, B. Expectativas del clínico
frente a la utilización de los test de resistencias. En: Clotet,
B., Ruiz, L., Martínez-Picado, J., Gatell, J.M., Menéndez-Arias,
L., Richman, D.D. (Eds.). Guía práctica para el manejo clínico
de las resistencias al VIH. Grupo E. Entheos, Madrid 1999; 269-274. 84. Clotet, B., Gatell, J.M. Introducción
de los test de resistencia al VIH en la práctica clínica.
En: Clotet, B., Ruiz, L., Martínez-Picado, J., Gatell, J.M., Menéndez-Arias,
L., Richman, D.D. (Eds.). Guía práctica para el manejo clínico
de las resistencias al VIH. Grupo E. Entheos. Madrid 1999; 15-28. 85. Ortiz de Lejarazu, R., Cisterna,
R., Eiros, J.M. y cols. Diagnóstico microbiológico de
la infección por VIH, 6B. En: Picazo, J.J. (Ed.). Procedimientos
en microbiología clínica. SEIMC, Madrid 1998. 86. Córdoba, J., Esteban, R.J.,
Garijo, R., Molina, J.M., Gobernado, M. Resistencias del VIH-1 y el
laboratorio clínico. Rev Esp Quimioterap 1999; 12: 310-316. 87. Innis, M., Gelfand, D. Optimization
of PCRs. En: Innis, M., Gelfand, D., Sninsky, J. y cols. (Eds.). PCR
Protocols. A Guide to Methods and Applications. Academic Press, Inc.,
San Diego 1990; 3-12. 88. Cline, J., Braham, J., Hogrefe, H.
PCR fidelity of Pfu DNA polymerase and other termostable DNA polymerases.
Nucl Acids Res 1996; 24: 3546-3550. 89. Richman, D. Implications of resistance
testing for management: Role in selecting an initial regimen and use of
resistance testing when changing therapy. En: Grupo E. Entheos. Guía
práctica para el manejo clínico de las resistencias al VIH.
Gráficas Marte, Madrid 1999; 135-144. 90. Bonhoeffer, S., May, R.M., Shaw,
G.M., Novak, M.A. Virus dynamics and drug therapy. Proc Natl Acad
Sci USA 1997; 94: 6971-6976. 91. Maschera, B., Furfine, E., Byair,
E.D. Analysis of resistance to human immunodeficiency virus type 1
protease inhibitors by using matched bacterial expression and proviral
infected vectors. J Virol 1995; 69: 5431-5436. 92. Geretti, A.M., Smith, M., Osner,
N. y cols. Prevalence of antiretroviral resistance in a South London
Cohort of treatment-naive HIV-1 infected patients. AIDS 2001; 15:
1082-1084. 93. Havlir, D.V., Hellmann, N.S., Petropoulus,
C.J. y cols. Drug susceptibility in HIV infection after viral rebound
in patients receiving indinavir-containing regimens. JAMA 2000; 283:
229-234. 94. Soriano, V., Ledesma, E. and the
Spanish Drug Resistance Panel. Second Spanish Consensus on the use
of drug resistance testing in clinical practice. AIDS Rev 2000; 2:
111-118. 95. Carpenter, C., Fischl, M., Hammer,
S. y cols. Antiretroviral therapy for HIV infection in 1998. JAMA
1998; 280: 78-86. 96. Little, S.J., Daar, E.S., D'Aquila,
R.T. y cols. Reduced antiretroviral drug susceptibility among patients
with primary HIV infection. JAMA 1999; 282: 1142-1149. 97. Shafer, R.W., Hertogs, K., Zolopa,
A.R. y cols. High degree of interlaboratory reproductibility of human
immunodeficiency type 1 protease and reverse transcriptase sequencing
of plasma samples from heavily treated patients. J Clin Microbiol
2001; 39: 1522-1529. 98. Hanna, G.J., D'Aquila, R.T. Clinical
use of genotypic and phenotypic drug resistance testing to monitor antiretroviral
chemotherapy. Clin Infect Dis 2001; 32: 774-782. 99. Gilbert, P.B., Hanna, G.J., De Gruttola,
V. y cols. Comparative analysis of HIV type 1 genotipic resistance
across antiretroviral trial treatment regimens. AIDS Res Hum Retroviruses
2000; 16: 1325-1336. 100. Kartsonis, N.A., D'Aquila, R.T. Clinical
monitoring of HIV-1 infection in the era of antiretroviral resistance
testing. Infect Dis Clin North Am 2000; 14: 879-899. 101. Hanna, G.J., D'Aquila, R.T. Antiretroviral
drug resistance in HIV-1. Curr Infect Dis Rep 1999; 1: 289-297. 102. Vandamme, A., Van Vaerenbergh, K.,
De Clerq, E. Anti-HIV drug combination strategies. Antiviral Chem
Chemother 1998; 9: 187-203. 103. Jiménez-Náchez, I.,
Soriano, V. Interactions of antiretroviral drugs. AIDS Rev 1999;
1: 116-123. 104. Markowitz, M. Resistance, fitness,
adherence and potency: Mapping the path to virologic failure. JAMA
2000; 283: 250-251. 105. Martínez-Picado, J., De Pasquale,
M.P., Kartsonis, N. y cols. Antiretroviral resistance during successful
therapy of HIV type 1 infection. Proc Natl Acad Sci USA 2000; 97:
10948-10953. 106. Chesney, M., Ickovics, J., for the
Recruitment, Adherence and Retention Committee of the ACTG. Adherence
to combination therapy in AIDS clinical trials. Anual Meeting of the
AIDS Clinical Trials Group, Washington, DC 1997. 107. Grupo de Estudio de Sida de la SEIMC,
Sociedad Española de Farmacia Hospitalaria y Secretaría
del Plan Nacional sobre el Sida. Recomendaciones para mejorar la adherencia
al tratamiento antirretrovírico. Ministerio de Sanidad y Consumo,
Madrid 1999. 108. Núñez, M., Rodríguez-Rosado,
R., Soriano, V. Intensification of antirretroviral therapy. AIDS
Res Hum Retroviruses 2001; 17: 499-506. 109. Verbiest, W., Brown, S., Cohen, S.
y cols. Prevalence of HIV-1 drug resistance in antiretroviral-naive
patients: A prospective study. AIDS 2001; 15: 647-650. 110. Powderly, W., Landay, A., Lederman,
M. Recovery of the immune system with antiretroviral therapy. The end
of opportunism? JAMA 1998; 280: 72-77. 111. Soriano, V., Miró, J.M., Guerrero,
A. Resistencias primarias a los antirretrovirales. Enferm Infecc
Microbiol Clin 2001; 19 (monogr.): 22-25. |