Transferencia génica trans-gram: el caso de las betalactamasas
J.C. Galán
Servicio de Microbiología, Hospital Ramón y Cajal, Madrid.
|
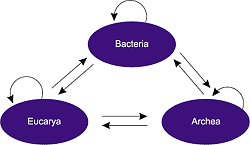
RESUMEN La transferencia horizontal de genes es un fenómeno que ha contribuido fuertemente en la evolución de los microorganismos. A pesar de la tendencia evolutiva al aislamiento genético, parece ser que la transferencia horizontal tiene lugar con una frecuencia relativamente alta, incluso entre especies muy separadas filogenéticamente. La adquisición de genes por las bacterias puede haberse acelerado en los últimos años debido a un incremento en sus necesidades adaptativas, particularmente por el uso de antibióticos. A la luz del análisis de las secuencias de aminoácidos se ha sugerido la transferencia de genes que codifican betalactamasas desde bacterias grampositivas a gramnegativas (transferencia trans-gram). Al realizar árboles filogenéticos con betalactamasas de diferentes orígenes encontramos que las BRO-1 y ACI-1, descritas en las bacterias gramnegativas Moraxella y Acidaminococcus, respectivamente, están anormalmente agrupadas con las betalactamasas de las bacterias grampositivas. En ambos casos la topología de la enzima (anclada a la membrana celular), la estructura del péptido señal y la región Shine-Dalgarno sugieren que esas enzimas tuvieron su origen en bacterias grampositivas. Por otra parte, los resultados siguen sin ser definitivos para la betalactamasa ROB-1 de Haemophilus. Palabras clave: Betalactamasa - BRO-1 - ACI-1 - Transferencia horizontal Trans-gram gene transfer: The case of b-lactamases SUMMARY Lateral or horizontal gene transfer is a phenomenon that has influenced the evolution of microorganisms. Despite the evolutionary trend toward genetic isolation, lateral transfer seems to take place relatively frequently, bridging the gap between very separate species. The acquisition of foreign genes may have accelerated in recent years because of the increase in the adaptive needs of bacteria, particularly through the use of antibiotics. Transfer of genes encoding ß-lactamases from Gram-positive to Gram-negative bacteria (trans-gram transfer) may be suggested on the basis of sequence analysis. We found that the sequences of the ß-lactamases BRO-1 and ACI-1, from the Gram-negative bacterial organisms Moraxella and Acidaminococcus, respectively, are abnormally placed among sequences from Gram-positive ß-lactamases in phylogenetic trees. In both cases, the topology of the enzyme (attached to the cellular membrane), the structure of the signal peptide, and the Shine-Dalgarno region suggest that these enzymes originated from Gram-positive organisms. Results remain inconclusive for Haemophilus ROB-1 ß-lactamase. Key words: Betalactamase - BRO-1 - ACI-1 - Horizontal gene transfer INTRODUCCIÓN La transferencia de genes entre especies bacterianas relacionadas y no relacionadas filogenéticamente es un proceso generalizado en el mundo microbiano. Se trata de uno de los mecanismos más importantes en la evolución de los genomas, implicado en la aparición de nuevos caracteres dentro de una especie (por ejemplo variantes más virulentas) o en la emergencia de nuevas especies (1-3). La transferencia horizontal de genes es un fenómeno universal que tiene lugar en y entre los tres dominios de la vida (4-8) (Fig. 1). A medida que más genomas sean secuenciados completamente, más precisa será la estimación cuantitativa de la importancia del flujo de información genética entre especies. Los datos analizados hasta la fecha permiten considerar que este fenómeno ha ocurrido y sigue ocurriendo con una apreciable frecuencia, y de manera constante hasta nuestros días (9, 10); así, se ha estimado que Escherichia coli reemplaza por intercambio horizontal 16 kb de su genoma cada millón de años (11).
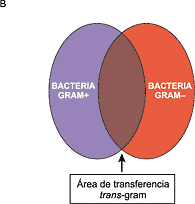
Figura 1. Representación esquemática de la universalidad de la transferencia horizontal de genes entre los dominios de la vida. Con algunas limitaciones, los dos criterios universalmente empleados para detectar la huella de la transferencia horizontal de genes son la existencia de un gen o secuencia génica de una composición nucleotídica anómala respecto a la del resto del genoma, y la localización incongruente de un gen o secuencia génica en el árbol filogenético del organismo (12). Cuando se sospeche cuál puede ser el microorganismo del que procede el DNA exógeno (que ha de compartir con el receptor, en algún momento, un ecosistema común), debería encontrarse la secuencia transferida en una elevada proporción de la población donadora, así como su rareza en el microorganismo receptor (13), e identificarse en el donador y el receptor una misma disposición génica en dicha secuencia (14). Empleando estos criterios, y aplicando programas informáticos de modelización, se ha establecido que la tasa de DNA exógeno que forma parte del genoma de una bacteria oscila entre el 1,5% en bacterias con nichos muy específicos (como Mycoplasma genitalium) y el 14,5% en bacterias ambientales y ubicuas (como Bacillus subtilis) (7, 14). Si sólo se considera la transferencia entre dominios o especies muy alejadas filogenéticamente los resultados son drásticamente inferiores: entre <0,5% y 1% en bacterias patógenas y aproximadamente el 3% en bacterias ambientales (14). ¿Cómo puede tener lugar la transferencia horizontal de genes entre especies bacterianas muy alejadas filogenéticamente, por ejemplo entre grampositivos y gramnegativos? Como es bien conocido, existen tres procedimientos de captación de DNA exógeno: transformación, transducción y conjugación. Los dos primeros parecen poco efectivos para transferir DNA entre especies poco relacionadas. Si bien la transformación puede ocurrir en la naturaleza en especies que son transformantes naturales (como Streptococcus pneumoniae o B. subtilis), ésta sólo tendría lugar en caso de que la divergencia génica entre el DNA del huésped y el DNA exógeno fuese inferior al 25%. En la transferencia mediada por fagos (transducción) se han descrito fagos capaces de replicarse tanto en grampositivos como en gramnegativos (15), pero esta observación es de carácter excepcional. El mecanismo más probable de transferencia genética entre especies poco relacionadas es el de la conjugación, ya que existe una serie de elementos genéticos conjugativos muy promiscuos (plásmidos y transposones), capaces de transferirse entre bacterias grampositivas y gramnegativas y mantenerse en ellas (17-20). TRANSFERENCIA GENÉTICA ENTRE BACTERIAS GRAMPOSITIVAS Y GRAMNEGATIVAS El estudio de la transferencia genética entre bacterias grampositivas y gramnegativas (transferencia trans-gram, según el término acuñado por Courvalin) (13) debería, en efecto, proporcionar las mejores claves para la comprensión del intercambio de genes entre especies poco relacionadas genéticamente, ya que la separación evolutiva de estos dos grupos bacterianos es la más profunda escisión del árbol filo genético de los procariotas (21). De acuerdo con el postulado de limitación de flujo génico entre organismos filogenéticamente separados (Fig. 2A), la transferencia genética trans-gram se considera generalmente un suceso de baja frecuencia; sin embargo, hoy sabemos que este flujo de información podría ser mayor de lo que inicialmente fue estimado y que es probable que exista una relación genética entre ambos grupos bacterianos (Fig. 2B). Esta zona de intercambio podría estar facilitada por la persistencia en la naturaleza de especies bacterianas ancestrales, con caracteres intermedios entre grampositivas y gramnegativas que facilitarían el flujo de información entre grupos o especies poco relacionadas filogenéticamente.
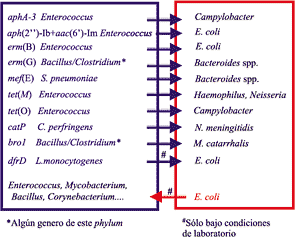
Figura 2. Representación esquemática del flujo de información genética entre especies bacterianas. A: El grosor de las flechas indica la mayor o menor frecuencia con que tales procesos tienen lugar. B: Transferencia genética entre bacterias grampositivas y gramnegativas. La transferencia de genes de resistencia antibiótica constituye un buen modelo para cuantificar la diseminación de un determinado gen, no sólo por ser un suceso fácilmente detectable sino por estar delimitado en el periodo de los últimos 50 o 60 años, cuando la presión antibiótica en el medio ha sido más agresiva. Las primeras ocasiones en que se comunican en la literatura transferencias trans-gram son los trabajos de Trieu-Cuot y cols., que detectaron la transferencia del gen aphA-3, que codifica para una enzima modificante de aminoglucósidos, desde Enterococcus o Staphylococcus a Campylobacter (22, 23). En la actualidad, para todos los grandes grupos de antimicrobianos se ha documentado o sugerido un proceso de transferencia trans-gram (Fig. 3).
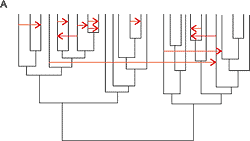
Figura 3. Ejemplos de genes de resistencia antibiótica que han sido transferidos desde bacterias grampositivas a otras gramnegativas (50-56). Aparentemente la transferencia trans-gram posee un fuerte carácter asimétrico. Como se observa en la Fig. 3, todos los casos descritos de transferencia trans-gram se refieren a transferencia desde bacterias grampositivas a bacterias gramnegativas. ¿Por qué es tan unidireccional esta transferencia? Aparentemente, el flujo de información genética entre bacterias grampositivas y gramnegativas podría ser bidireccional y simétrico, pero no es así en la naturaleza. La respuesta es que los genes de las bacterias grampositivas se expresan fácilmente en las gramnegativas, mientras que lo contrario es más difícil, debido a que la maquinaria de la traducción en las bacterias grampositivas es más restrictiva (24). Desde un punto de vista evolutivo, la permanencia de un gen en un nuevo huésped depende de su estabilidad y expresión (25); probablemente, si el gen proporciona una ventaja adaptativa la permanencia se facilita. Se conoce poco sobre el coste biológico relativo de la presencia de un gen de grampositivos en gramnegativos, y viceversa. Cabe imaginar que, incluso si un gen de una bacteria gramnegativa pudiese ser adaptativamente útil en una bacteria grampositiva, el coste biológico de su mantenimiento pudiese llegar a ser excesivo en periodos de bajo grado de selección. La direccionalidad de la transferencia trans-gram se explicaría si el coste de mantener un gen trans-gram fuese superior para las bacterias grampositivas que para las gramnegativas. Incluso con bajo coste, si el gen no pudiera expresarse, es de esperar que en sucesivas generaciones podrían acumularse mutaciones deletéreas en dicho gen, convirtiéndose progresivamente en la denominada "basura génica" que sería finalmente eliminada (teoría reduccionista de la evolución). El grado de expresión de un gen adquirido por transferencia horizontal depende de la eficiencia con que la RNA polimerasa reconoce las secuencias promotoras, y los ribosomas la secuencia Shine-Dalgarno del RNAm. Los trabajos de Kasak y cols. revelan que al introducir por manipulación genética en un organismo un gen que carece de secuencia promotora, después de tres días en fase estacionaria aparecen, bajo condiciones de selección, mutaciones en la región por delante del gen introducido que generan secuencias promotoras (26). De la misma manera, cuando se introducen genes cuya región promotora no es reconocida eficientemente por la RNA polimerasa del nuevo huésped, a los dos o tres días de permanencia del cultivo en fase estacionaria aparecen mutaciones en la secuencia promotora, lo que permite un más fácil reconocimiento por la RNA polimerasa, incrementando su expresión (27). Por lo tanto, no parece que la eficacia con que la RNA polimerasa reconoce las posibles secuencias promotoras sea la causa principal de que la transferencia trans-gram sea unidireccional. Por el contrario, los trabajos de McLaughlin y cols. (24) demuestran que la mayoría de los sitios de iniciación del RNAm de las bacterias gramnegativas no son reconocidos eficientemente por los ribosomas de las bacterias grampositivas, debido a que estos ribosomas requieren una secuencia Shine- Dalgarno más extensa y fuertemente complementaria del extremo 3' del 16S RNAr, tolerando menor variabilidad que la observada en los sitios de unión al ribosoma en las bacterias gramnegativas. Esta diferencia está relacionada con la reducida dependencia del factor de iniciación IF3 en el comienzo de la traducción en las bacterias grampositivas (28). TRANSFERENCIA TRANS-GRAM DE BETALACTAMASAS El fenómeno de transferencia trans-gram se ha podido documentar en algunos casos en relación con genes de resistencia a antibióticos betalactámicos. Las betalactamasas BRO de Moraxella catarrhalis (BRO-1, BRO-2 y BRO-3) fueron las primeras que se describieron en una bacteria gramnegativa cuyo origen podría estar en una bacteria grampositiva (29). De hecho, la secuencia de la betalactamasa BRO-1 tiene una posición incongruente en árboles filogenéticos (Fig. 4), agrupándose con las betalactamasas de las bacterias grampositivas (30), lo que sugiere fuertemente su origen en este grupo de microorganismos. De lo que no cabe duda es del éxito adaptativo de estas enzimas para la supervivencia de Moraxella en un ambiente bajo alta presión antibiótica.
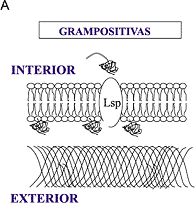
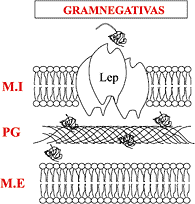
Figura 4. Árbol filogenético, según criterio de parsimonia, de 46 betalactamasas de diferentes orígenes. En azul se indican las betalactamasas descritas en bacterias grampositivas y en rojo las descritas en gramnegativas. (Adaptada de ref. 30 y modificada para incluir ACI-1 según ref. 38.) En 1977 se describió la primera cepa de M. catarrhalis resistente a los betalactámicos por producción de una betalactamasa (31). A mediados de la década de 1990, el 90% de las cepas eran ya productoras de la enzima (32, 33). Esta brusca subida representa uno de los más rápidos incrementos en la prevalencia de cualquier betalactamasa conocida en una especie bacteriana. La secuenciación de los genes bro que codifican para la betalactamasa reveló un contenido G+C del 31%, muy diferente del esperado para genes cromosómicos de M. catarrhalis (34). La ausencia, en las cepas sensibles, de regiones homólogas a los genes bro, descarta la posibilidad de sucesos mutacionales que hubiesen podido dar lugar a betalactamasas a partir de otros genes cromosómicos que habrían evolucionado hacia nuevas funciones en el DNA preexistente (35). Todo ello sugiere que los genes bro han llegado a Moraxella por un proceso de transferencia horizontal (probablemente por transformación de DNA cromosómico de una especie grampositiva, ya que M. catarrhalis es competente natural y no se encuentran elementos móviles asociados a bro). Lo más probable es que este proceso haya ocurrido recientemente, debido al bajo polimorfismo genético en el gen bro en comparación con los genes adyacentes (36), y que haya sido facilitado por el consumo de antimicrobianos. Como se muestra en la Fig. 4, otras dos betalactamasas encontradas en bacterias gramnegativas son sospechosas de proceder evolutivamente de bacterias grampositivas: las betalactamasas ROB-1 de Haemophilus influenzae y ACI-1 de Acidaminococcus fermentans. Ambas se localizan en una posición incongruente en el árbol filogenético de las betalactamasas, con un mayor grado de homología con las betalactamasas de las bacterias grampositivas (63% y 45%, respectivamente) que con las de las gramnegativas (24%). Por tanto, podrían haber emergido en Haemophilus y Acidaminococcus a partir de una transferencia trans-gram (37, 38). En el caso de la betalactamasa ROB-1, Livrelli y cols. (39) llegan a una conclusión similar al observar en la secuencia proteica el doblete Asn-Asp en las posiciones 245-246, que parece exclusivo, según los autores, de las betalactamasas encontradas en bacterias grampositivas. No se ha hallado el gen blaROB-1 fuera de miembros de la familia Pasteurellaceae (Pasteurella, Actinobacillus y Haemophilus, con más del 99% de identidad génica) (40), y el análisis de los perfiles plasmídicos de los diferentes plásmidos que portan el gen blaROB-1 refleja un alto grado de identidad entre sí, lo que sugiere que la adquisición de este gen por el plásmido, o de un plásmido ancestral que portara el gen de resistencia, ocurrió recientemente (41, 42). Por otra parte, en la betalactamasa ACI-1 ya mencionada, recientemente descrita en un coco gramnegativo anaerobio estricto, hemos sugerido (38) un proceso de transferencia trans-gram a la luz del análisis de su secuencia nucleotídica (un contenido G+C del 42% respecto al 56% del DNA genómico) y proteica (presencia del doblete Asn-Asp en las posiciones 245-246). Se sugiere que esta transferencia debe ser igualmente reciente, ya que la diferencia en el contenido G+C en la tercera posición del triplete es aún mas acentuada respecto a la de los genes cromosómicos de la bacteria receptora (35% respecto del 63%). Recordemos que en las secuencias de DNA que codifican proteínas las diferencias en G+C tienden a anularse, por cambios selectivos en la tercera posición del triplete (43). La localización celular de las betalactamasas encontradas en las bacterias grampositivas respecto de las halladas en las bacterias gramnegativas nos permite apoyar la hipótesis del origen evolutivo de BRO-1, ROB-1 y ACI-1 en bacterias grampositivas. Las betalactamasas de las bacterias grampositivas son verdaderas lipoproteínas ancladas a la membrana (44). Esta particularidad evita, en gran medida, que estas betalactamasas puedan diluirse por difusión libre al medio circundante, lo que disminuiría su capacidad de proteger a la bacteria del efecto letal de los betalactámicos (45). Esta característica no es necesaria en las betalactamasas de las bacterias gramnegativas, ya que la enzima madura queda retenida en el espacio periplásmico (Fig. 5A). Esta diferente localización de los dos grandes grupos de betalactamasas es el reflejo de un diferente procesamiento del péptido señal que permite la salida de la proteína del citoplasma bacteriano a través de la membrana. En el caso de las betalactamasas de las bacterias gramnegativas, el péptido señal es reconocido por una peptidasa de señal tipo I (LepB), que reconoce la secuencia (A/V)-x-A-x (46). Por el contrario, en las betalactamasas de las bacterias grampositivas el péptido señal es reconocido por una peptidasa de señal tipo II (LspA), embebida en la membrana citoplásmica, que reconoce una secuencia señal tipo L-(A/S/T/I)-(A/G)-C-(G/S), donde Cys es el primer aminoácido de la proteína madura (47), y que queda unida a los lípidos de la membrana citoplásmica (Fig. 5B). Pues bien, en el hipotético péptido señal de las betalactamasas BRO-1, ROB-1 y ACI-1 se encuentran secuencias reconocidas por la peptidasa de señal tipo II, si bien en el caso de ROB-1 de H. influenzae también existe una secuencia que puede ser reconocida por la peptidasa de señal tipo I (Fig. 5C).
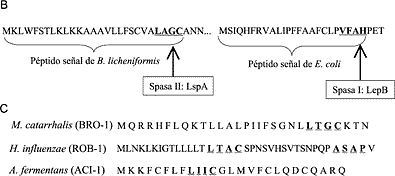
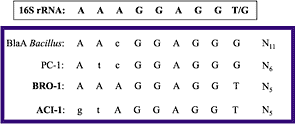
Figura 5. Diferente procesamiento de las betalactamasas de diferentes orígenes. A: Distinta localización celular de las betalactamasas procedentes de bacterias grampositivas y gramnegativas. B: Reconocimiento del péptido señal de la betalactamasa de Bacillus licheniformis y E. coli. C: Hipotéticos péptidos señales de las posibles betalactamasas trans-gram; en negrita y subrayado se destacan las posibles secuencias que son reconocidas por la peptidasa de señal tipo I o tipo II. La betalactamasa BRO-1 presenta la secuencia señal LTGC, encontrada también en otras lipoproteínas de bacterias grampositivas (como YrpE). Mediante microscopía electrónica y empleo de anticuerpos anti-BRO, Bootsma y cols. (29) han podido demostrar que cuando BRO-1 se expresa en E. coli un 45% de la actividad enzimática se encuentra unida a la cara interna de la membrana externa (mientras que empleando TEM-1 como control tan sólo el 0,2% aparece fijado a la membrana), siendo Cys el primer aminoácido de la proteína madura. En este mismo sentido, la betalactamasa ACI-1 presenta la secuencia señal LIIC, también encontrada en otras lipoproteínas de bacterias grampositivas (como YoaO). Los trabajos sobre fraccionamiento celular (48) de la enzima ACI-1 expresada en E. coli, realizados en nuestro laboratorio, revelan que la mayor actividad enzimática aparece en la fracción de membrana, seguida de la fracción periplásmica y finalmente de la fracción citoplásmica (49). Estos datos sugieren fuertemente que ambas proteínas, BRO-1 y ACI-1, en gramnegativos como Moraxella y Acidaminococcus, podrían tener su origen en bacterias grampositivas. El hecho de que no exista ningún estudio sobre la afinidad de la betalactamasa ROB-1 por la membrana bacteriana, y la presencia de posibles sitios de reconocimiento para peptidasas de señal tipo I (ASAP) y II (LTAC), hacen que por el momento no sea definitivo el argumento de que la aparición de ROB-1 en Haemophilus pueda constituir un nuevo ejemplo de transferencia trans-gram. Como ya se ha comentado, la principal restricción para la permanencia en un huésped bacteriano de material génico procedente de otro alejado filogenéticamente es la limitación en la expresión, en el nuevo huésped, del gen o genes transferidos. Dicha expresión depende, en las bacterias grampositivas, de la presencia de una extensa y conservada secuencia Shine-Dalgarno en el RNAm, por la cual los ribosomas deben mostrar una buena afinidad para que la expresión del gen transferido sea la adecuada. En este sentido, el hallazgo de sitios de unión de los ribosomas altamente conservados en las betalactamasas BRO-1, ROB-1 y ACI-1, sospechosas de proceder de bacterias grampositivas, apoyaría la posibilidad de una expresión correcta en bacterias gramnegativas. Como se muestra en la Fig. 6, el sitio de unión del ribosoma en los genes que codifican para betalactamasas de bacterias grampositivas, como blaPC-1 o blablaA, está más conservado que en las betalactamasas de las bacterias gramnegativas, como blaTEM-1, blaSHV-1 o blaPER-1. Hemos visto que las secuencias Shine-Dalgarno de las betalactamasas BRO-1 y ACI-1 están muy conservadas, como ocurre con las betalactamasas de las bacterias grampositivas. Sin embargo, ROB-1, como las betalactamasas de las bacterias gramnegativas, no presenta una secuencia Shine-Dalgarno muy conservada, y ello sería un criterio en contra de su origen en bacterias grampositivas. Pero podría sugerirse que la adquisición de dicho gen haya ocurrido hace mucho tiempo y la secuencia Shine-Dalgarno que actualmente aparece por delante del gen blaROB-1 fuera el resultado de una óptima adaptación a los ribosomas de H. influenzae. Esta posibilidad no parece probable, ya que la secuenciación del gen blaROB-1 de diferentes clones revela un escaso polimorfismo genético, lo que sugiere que el gen fue adquirido recientemente (41, 42).
Figura 6. Secuencias Shine-Dalgarno de betalactamasas de bacterias grampositivas y gramnegativas. En letras minúsculas se identifican las bases nucleotídicas que no corresponden con la secuencia consenso complementaria de extremo 3' del 16S RNAr. CONCLUSIONES La transferencia horizontal de genes es un proceso generalizado y constante que sigue ocurriendo en la actualidad, hasta tal punto que podría considerarse que las especies actuales son el resultado último del proceso de transferencia horizontal de genes (1). Dentro de este proceso universal, la transferencia entre especies muy alejadas también ocurre con una frecuencia superior a lo que inicialmente se había considerado. Se ha documentado transferencia trans-gram de una variedad de genes, especialmente genes de resistencia antibiótica, seguramente debido a la facilidad para su detección y al hecho de ser un fenómeno reciente, determinado por el consumo generalizado de antibióticos. Los procesos de transferencia trans-gram de genes de virulencia o genes de rutas metabólicas puede haberse estado desarrollando desde hace mucho más tiempo; de esta forma, habiéndose adaptado ya las secuencias adquiridas a sus nuevos huéspedes, la dificultad de detectar su existencia es mucho mayor (43). La transferencia trans-gram de genes que codifican para betalactamasas de bacterias grampositivas a bacterias gramnegativas ya se había sugerido previamente, basándose en datos de homología de secuencia. Sin embargo, según lo aquí presentado, aplicando varios criterios, como a) una posición incongruente de la secuencia proteica en los árboles de filogenia, b) contenido G+C significativamente diferente entre el putativo gen transferido y otros genes cromosómicos, c) localización celular de la enzima, principalmente en cuanto a su unión a la membrana bacteriana y los resultados del análisis del péptido señal, y d) análisis del sitio de unión de sus RNAm al ribosoma, se puede concluir que las betalactamasas BRO-1 y ACI-1 probablemente tuvieron su origen en bacterias grampositivas, mientras que en el caso de ROB-1 se requieren nuevos estudios antes de concluir que pueda tratarse de una betalactamasa trans-gram. AGRADECIMIENTOS El autor agradece la orientación y discusión del tema de este artículo con el Dr. D. Fernando Baquero. Para el desarrollo de este trabajo el autor ha recibido ayuda de una Beca BEFI 98/9060 y del Proyecto DEAR QLK2-CT2001-00-873 de la Unión Europea. BIBLIOGRAFÍA 1. De la Cruz, F., Davies, J. Horizontal gene transfer and the origin of species: Lessons from bacteria. Trends Microbiol 2000; 8: 128-133. 2. Hinnebush, B.J., Rudolph, A.E., Cherepanov, P., Dixon, J.E., Schwan, T.G., Forsberg, A. Role of yersinia murine toxin in survival of Yersinia pestis in the midgut of the flea vector. Science 2002; 296: 733-735. 3. Ohnishi, M., Kurokawa, K., Hayashi, T. Diversification of Escherichia coli genomes: Are bacteriophages the major contributors? Trends Microbiol 2001; 9: 481-485. 4. Ochman, H., Lawrwnce, J.G., Groisman, E.A. Lateral gene transfer and natural of bacterial innovation. Nature 2000; 405: 299-304. 5. Berg, O.G., Kurland, C.G. Why mitochondrial genes are most often found in nuclei? Mol Biol Evol 2000; 17: 951-961. 6. Nelson, K.E., Clayton, R.A., Gill, S.R. y cols. Evidence for lateral gene transfer between Archaea and Bacteria from genome sequence of Thermotoga maritima. Nature 1999; 399: 323-329. 7. García-Vallvé, S., Romeu, A., Palau, J. Horizontal gene transfer of glycosyl hydrolases of the rumen fungi. Mol Biol Evol 2000; 17: 352-361. 8. Sicot, F.X., Mesnege, M., Masselot, M. y cols. Molecular adaptation to an extreme enviroment: Origin of the thermal stability of the pompeii worm collagen. J Mol Biol 2000; 302: 811-820. 9. Jain, R., Rivera, M.C., Lake, J.A. Horizontal gene transfer among genomes: The complexity hypothesis. Proc Natl Acad Sci USA 1999; 96: 3801-3806. 10. Hendrix, R.W., Smith, M.C.M., Burns, R.N., Ford, M.E., Hatfull, G.F. Evolutionary relationships among diverse bacteriophages and prophages: All the world's a phage. Proc Natl Acad Sci USA 1999; 96: 2192-2197. 11. Lawrence, J.G., Ochman, H. Molecular archaeology of the Escherichia coli genome. Proc Natl Acad Sci USA 1998; 95: 9413-9417. 12. Lawrence, J.G., Ochman, H. Reconciling the many faces of lateral gene transfer. Trends Microbiol 2002; 10: 1-4. 13. Courvalin, P. Transfer of antibiotic resistance gene between grampositive and gramnegative bacteria. Antimicrob Agents Chemother 1994; 38: 1447-1451. 14. Koonin, E.V., Makarova, K.S., Gravin, L. Horizontal gene transfer in prokaryotes: Quantification and classification. Annu Rev Microbiol 2001; 55: 709-742. 15. Álvarez, M.A., Herrero, M., Suárez, J.E. The site-specific recombination system of the Lactobacillus species bacteriophage A2 integrates in grampositive and gramnegative bacteria. Virology 1998; 250: 185-193. 16. Rawling, D.E., Tietze, E. Comparative biology of IncQ and IncQ-like plasmids. Microbiol Mol Biol Rev 2001; 65: 481-496. 17. Szpirer, C.Y., Faelen, M., Couturier, M. Mobilization function of the pBHR1 plasmid, a derivative of the broad-host-range plasmid pBBR1. J Bacteriol 2001; 183: 2101-2110. 18. Charpentier, E., Gerbaud, G., Courvalin, P. Conjugative mobilization of the rolling-circle plasmid pIP823 from Listeria monocytogenes BM4293 among grampositive and gramnegative. J Bacteriol 1999; 181: 3368-3374. 19. Bertram, J., Strätz, M., Dürre, P. Natural transfer of conjugative transposon Tn916 between grampositive and gramnegative bacteria. J Bacteriol 1991; 173: 443-448. 20. Poyart, C., Celli, J., Trieu-Cuot, P. Conjugative transposition of Tn916-related elements from Enterococcus faecalis to Escherichia coli and Pseudomonas fluorescens. Antimicrob Agents Chemother 1995; 39: 500-506. 21. Gupta, R.S. Protein phylogenies and signature sequences: A reappraisal of evolutionary relationships among archaebacteria, eubacteria, and eukaryotes. Microbiol Mol Biol Rev 1998; 62: 1435-1491. 22. Trieu-Cuot, P., Gerbaud, G., Lambert, T., Courvalin, P. In vitro transfer of genetic information between Grampositive and Gramnegative bacteria. EMBO J 1985a; 4: 3538-3587. 23. Trieu-Cuot, P., Klier, A., Courvalin, P. DNA sequences specifying the transcription of the streptococcal kanamycin resistance gene in Escherichia coli and Bacillus subtilis. Mol Gen Genet 1985b; 198: 348-352. 24. McLaughlin, J.R., Murray, C.L., Rabinowitz, J.C. Unique features in the ribosome binding site sequence of the grampositive Staphylococcus aureus b-lactamase gene. J Biol Chem 1981; 21: 11283-11291. 25. Mazodier, P., Davies, J. Gene transfer between distantly related bacteria. Annu Rev Genet 1991; 25: 147-171. 26. Kasak, L., Hörak, R., Kivisaar, M. Promoter-creating mutations in Pseudomonas putida: A model system for the study of mutation in starving bacteria. Proc Natl Acad Sci USA 1997; 94: 3134-3139. 27. Dabizzi, S., Ammannato, S., Fani, R. Expression of horizontally transferred gene clusters: Activation by promoter-generating mutations. Res Microbiol 2001; 152: 539-549. 28. McLaughlin, J.R., Murray, C.L., Rabinowitz, J.C. Initiation factor-independent translation of mRNAs from grampositive bacteria. Proc Natl Acad Sci USA 1981; 78: 4912-4916. 29. Bootsma, H.J., Aerts, P.C., Posthuma, G. y cols. Moraxella (Branhamella) catarrhalis BRO b-lactamase: A lipoprotein of grampositive origin? J Bacteriol 1999; 181: 5090-5093. 30. Massova, I., Mobashery, S. Kinship and diversification of bacterial penicillin-binding proteins and b-lactamases. Antimicrob Agents Chemother 1998; 42: 1-17. 31. Percival, A., Corkill, J.E., Rowlands, J., Sykes, R.B. Pathogenicity of a b-lactamase production by Branhamella (Neisseria) catarrhalis. Lancet 1977; ii: 1175. 32. Doern, G.V., Brueggemann, A.B., Pierce, G., Hogan, T., Holley, H.P., Jr., Rauch, A. Prevalence of antimicrobial resistance among 723 outpatient clinical isolates of Moraxella catarrhalis in United States in 1994 and 1995: Results of a 30-center national surveillance study. Antimicrob Agents Chemother 1996; 40: 2884-2886. 33. Schmitz, F.J., Beeck, A., Perdikouli, M. y cols. Production of BRO b-lactamases and resistance to complement in European Moraxella catarrhalis isolates. J Clin Microbiol 2002; 40: 1546-1548. 34. Bootsma, H.J., van Dijk, H., Verhoef, J., Fleer, A., Mooi, F.R.. Molecular characterization of the BRO b-lactamase Moraxella (Branhamella) catarrhalis. Antimicrob Agents Chemother 1996; 40: 966-972. 35. Walker, E.S., Levy, F. Genetic trends in a population evolving antibiotic resistance. Evolution 2001; 55: 1110-1122. 36. Bootsma, H.J., van Dijk, H., Vauterin P., Verhoef, J., Mooi, F.R. Genenis of BRO b-lactamase-producing Moraxella catarrhalis: Evidence for transformation-mediated horizontal transfer. Mol Microbiol 2000; 36: 93-104. 37. Juteau, J.M., Levesque, R. Sequence analysis and evolutionary perspective in ROB-1 b-lactamase. Antimicrob Agents Chemother 1990; 34: 1354-1359. 38. Galán, J.C., Reig, M., Navas, A., Baquero, F., Blázquez J. ACI-1 from Acidaminococcus fermentans: Characterization of the first b-lactamase in anaerobic cocci. Antimicrob Agents Chemother 2000; 44: 3144-3149. 39. Livrelli, V., Peduzzi, J., Joly, B. Sequence and molecular characterization of the ROB-1 b-lactamase gene from Pasteurella haemolytica. Antimicrob Agents Chemother 1991; 35: 242-251. 40. Chang, Y.F., Shi, J., Shin, S.J., Lein, D.H. Sequence analysis of the ROB-1 b-lactamase gene from Actinobacillus pleuropneumoniae. Vet Microbiol 1992; 32: 319-325. 41. Chang, Y.F., Ma, D.P., Bai, H.Q. y cols. Characterization of plasmids with antimicrobial resistant genes in Pasteurella haemolytica A1. DNA Seq 1992; 3: 89-97. 42. Azad, A.K., Coote, J.G., Parton, R. Distinct plasmid profiles of Pasteurella haemolytica serotypes and the characterization and amplification in Escherichia coli of ampicillin-resistance plasmids encoding ROB-1 b-lactamase. J Gen Microbiol 1992; 138: 1185-1196. 43. Lawrence, J.G., Ochman, H. Amelioration of bacterial genomes: Rates of change and exchange. J Mol Evol 1997; 44: 383-397. 44. Nielsen, J.B., Lampen, J.O. Membrane-bound penicillinase in grampositive bacteria. J Biol Chem 1982a; 25: 4490-4495. 45. Nielsen, J.B., Lampen, J.O. Glyceride-cysteine lipoproteins and secretion by gram-positive bacteria. J Bacteriol 1982b; 152: 315-322. 46. Palzkill, T., Le, Q.Q., Wong, A., Botstein, D. Selection of functional signal peptide cleavage site from a library of random sequence. J Bacteriol 1994; 176: 563-568. 47. Yamamoto, S., Lampen, J.O. Membrane penicillinase of Bacillus licheniformis 749/C: Sequence and possible repeated tetrapeptide structure of thr phospolipopeptide region. Proc Natl Acad Sci USA 1976; 73: 1457-1461. 48. Fraipont, C., Adam, M., Nguyen-Disteche, M. y cols. Engineering and overexpression of periplasmic form of the penicillin-binding protein 3 of Escherichia coli. Biochem J 1994; 298: 189-195. 49. Galán, J.C. Transferencia transgrámica de b-lactamasas. XVIII Congreso de la Sociedad Española de Microbiología, Alicante 2001; 14S. 50. Brisson-Noël, A., Arthur, M., Courvalin, P. Evidence for natural gene transfer from grampositive cocci to Escherichia coli. J Bacteriol 1988; 170: 1739-1745. 51. Wang, J., Shoemaker, N.B., Wang, G.R., Salyers, A.A. Characterization of a Bacteroides mobilizable transposon, NBU2, which carries a functional lincomycin resistance gene. J Bacteriol 2000; 182: 3559-3571. 52. Cooper, A.J., Shoemaker, N.B., Salyers, A.A. The erythromycin resistance gene from the Bacteroides conjugal transposon Tcr Emr 7853 is nearly identical to ermG from Bacillus sphericus. Antimicrob Agents Chemother 1996; 40: 506-508. 53. Galimand, M., Gerbaud,G., Guibourdenche, M., Riou, J.Y., Courvalin, P. High-level choramphenicol resistance in Neisseria meningitidis. N Engl J Med 1998; 339: 868-974. 54. Roberts, M.C. Characterization of the tetM determinants in urogenital and respiratory bacteria. Antimicrob Agents Chemother 1990; 34: 476-478. 55. Chow, J.W., Kalk, V., Kao, S.J. y cols. Aminoglycoside resistance gene aph(2'')-Ib and aac(6')-Im detected together in strain of both Escherichia coli and Enterococcus faecalis. Antimicrob Agents Chemother 2001; 45: 2691-2694. 56. Gormley, E.P., Davies, J. Transfer of plasmid RSF1010 by conjugation from Escherichia coli to Streptomyces lividans and Mycobacterium smegmatis. J Bacteriol 1991; 173: 6705-6708. Correspondencia: J.C. Galán, Servicio de Microbiología, Hospital Ramón y Cajal, Carretera de Colmenar km 9,100, 28035 Madrid. Fax: 91-3368809. e-mail: [email protected] |